Konzentration auf die Mission der Gruppe
Das SIB Clinical Bioinformatics unter der Leitung von Valérie Barbié und Aitana Neves bietet Fachwissen und Unterstützung bei der Organisation, Analyse und Interpretation von Patienten- und Gesundheitsdaten (z. B. Omics-Daten) und wandelt diese in klinisch verwertbare Informationen für medizinisches Fachpersonal um , um eine optimale Patientenversorgung zu gewährleisten. Entdecken Sie unten aktuelle Beispiele unserer Arbeit sowie unsere Fachkompetenz.
Die Gruppe leistet insbesondere einen Beitrag zu:
- Der Erfüllung von Branchenanforderungen durch Innovationsförderung und die Nutzung von Fachwissen in den Bereichen KI, Softwareentwicklung und Qualitätskontrolle; der Einrichtung modernster Diagnosetools für Schweizer Krankenhäuser und Kliniken;
- Entwicklung nationaler und internationaler Kooperationsplattformen zur Unterstützung der klinischen Forschung, Überwachung und Diagnostik;
- Bereitstellung von Training im Bereich klinische Bioinformatik in der ganzen Schweiz.
Auf die Bedürfnisse der Industrie eingehen
Die Gruppe arbeitet mit der Biotech- oder Pharmaindustrie zusammen, um neue Bioinformatik-Tools in verschiedenen Bereichen zu entwickeln, z. B. Bildgebung oder Genotypisierung. Aktuelle Beispiele sind:
Mit dem räumlichen Biologieunternehmen Lunaphore ermöglichte ein erster Innosuisse-Zuschuss eine Zusammenarbeit mit den Genfer Universitätskliniken. Ziel war es, Lösungen für maschinelles Lernen zu entwickeln, um die Analyse molekularer Bildgebungsdaten zur Charakterisierung von Tumormikroumgebungen zu unterstützen (Weiter lesen). Im Jahr 2023 begann dank einer zweiten Innosuisse-Förderung eine neue Zusammenarbeit zur Entwicklung KI-basierter Assay-Entwicklungstools, um den automatisierten Ansatz der COMET™-Plattform von Lunaphore zu verbessern und die Einführung der räumlichen Biologie in Forschungsprojekten weiter zu beschleunigen.
Weiterlesen über die Zusammenarbeit zwischen Lunaphore und SIB
- Für ein Schweizer Pharmaunternehmen Entwicklung einer Software zur Überwachung der genetischen Stabilität (Abwesenheit unerwünschter Mutationen) von Bakterienstämmen unter Verwendung von mikrofluidischen DNA-Chips während des gesamten Forschungs- und Herstellungsprozesses von Arzneimitteln.
- Kommerzialisierung von Melanie, der Standardsoftware von Das SIB zur Analyse von 2D-Elektrophorese-Gel- und Blot-Bildern. Ihre Anwendungsbereiche reichen von der Unterstützung bei der Entdeckung von Proteinbiomarkern und der Überwachung der Anpassung von Organismen an ihre Umgebung bis hin zur Entwicklung von Diagnosetests, der Qualitätskontrolle von Lebensmittelproben und der Entwicklung von HCP-ELISA-Tests (Auswahl und Validierung von Immunoassay-Reagenzien).
- Partner des Human Lean Diagnostics (HLD) -Hubs von Microcity (Innovationszentrum in Neuenburg), dessen Ziel es ist, die medizinische Diagnostik und HealthTech durch die Förderung der Zusammenarbeit und die Nutzung des kantonalen Know-hows im Bereich Mikrotechnologie zu unterstützen .
Modernste Diagnosegeräte für Krankenhäuser
Durch Partnerschaften mit Krankenhäusern entwickelt die Gruppe Sample-to-Report-Diagnosetools, die eine Optimierung und Automatisierung der Bioinformatik-Analyse von Patientendaten ermöglichen. Aktuelle Beispiele:
Entwicklung des Sample-to-Report-Tools OncoBench® in Zusammenarbeit mit den Genfer Universitätskliniken (HUG). Es wird seit 2016 von der Molekularpathologie der HUG für die Verwaltung und Analyse von NGS-Patientendaten in der routinemäßigen Krebsdiagnostik verwendet (derzeit in Version 5). Diese sichere webbasierte Plattform rationalisiert und automatisiert die komplexen molekularen Analysen, die zur Diagnose von Krebs führen, und gewährleistet gleichzeitig den Schutz der Patientendaten und die Einhaltung der Qualitätssicherungsstandards des Krankenhauses. Diese Art von Plattform kann für die molekulare Pathologiediagnostik in pathologischen Labors eingesetzt werden.
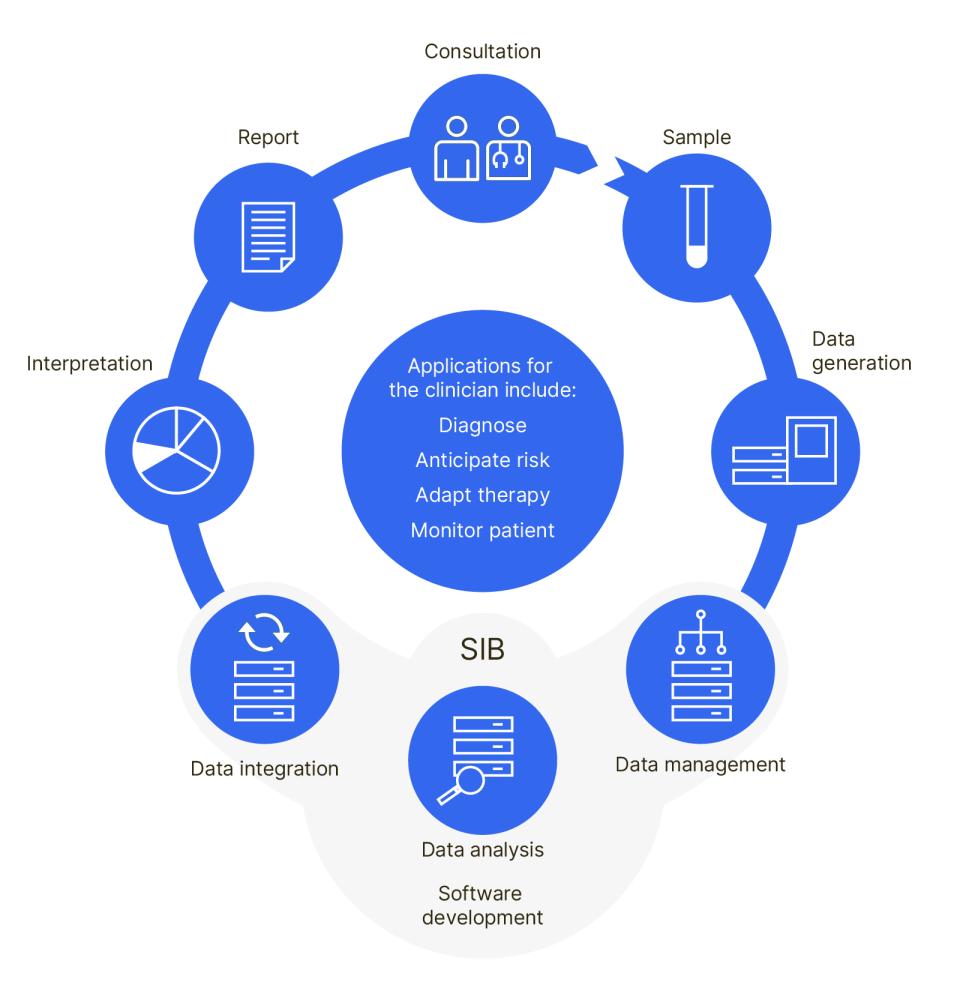
Beispiel für einen Datenfluss in der personalisierten Medizin, unterstützt durch das Fachwissen des SIB:
Unterstützung der landesweiten klinischen Forschung und Überwachung
Die Gruppe entwickelt nationale Kooperationslösungen zur Unterstützung der Omics-Forschung, der klinischen Diagnostik und der Pathogenüberwachung. Letzteres geschieht über das SIB-Zentrum für Pathogen-Bioinformatik unter der Leitung von Aitana Neves. Aktuelle Beispiele sind
- Gemeinsam mit mehreren Krankenhäusern und veterinärmedizinischen Einrichtungen leitet sie die Schweizer Plattform zur Überwachung von Krankheitserregern (SPSP), die nationale One-Health-Genomplattform zur Unterstützung der Pathogenüberwachung und -forschung. Zu den Herausforderungen gehören die Sicherstellung der Datenkonformität mit den Standards des Swiss Personalized Health Network (SPHN), die Durchführung bioinformatischer Analysen und die Verwaltung des kontrollierten Datenzugriffs für Nutzer wie klinische Mikrobiologielabore, Forscher und Gesundheitsbehörden. SPSP ist eine Ressource des SIB und wird vom Staatssekretariat für Bildung, Forschung und Innovation (SBFI) und dem Bundesamt für Gesundheit(BAG) finanziert.
- Mitleitung des Aufbaus der Plattform SwissGenVar, einer landesweiten Initiative, die erstmals alle wichtigen akademischen Einrichtungen für medizinische Genetik in der Schweiz miteinander verbindet. Ziel ist die Integration von diagnostisch relevanten Interpretationen genetischer Varianten und hochwertigen klinischen Daten (ursprünglich finanziert durch SPHN).
Bereitstellung von Training im Bereich klinische Bioinformatik in der ganzen Schweiz
Das Training in klinischer Bioinformatik für klinische Fachkräfte trägt zu einer verbesserten Kommunikation zwischen Experten verschiedener Disziplinen und zu einer besseren Nutzung dieser neuen Technologien im Alltag bei:
- A Certificate of Advanced Studies (CAS) in personalisierter molekularer Onkologie wird seit 2018 von der Gruppe in Zusammenarbeit mit den Universitätsspitäler Basel und Lausanne sowie der Universität Basel koordiniert. Dieses in der Schweiz einzigartige multidisziplinäre und standortübergreifende Programm zielt darauf ab, die nächste Generation von Fachleuten mit unterschiedlichem Hintergrund in diesem sich rasch entwickelnden Bereich auszubilden.
Unser Fachwissen
Für die Projekte, an denen wir gearbeitet haben, nutzen wir ein breites Spektrum an Know-how, darunter:
- Projekt- und Produktmanagement;
- Geschäftsanalyse;
- Wissenschaftliche Berechnungen und Analysen;
- Datenmanagement;
- Bioinformatik-Analyse;
- Maschinelles Lernen und Signal-/Bildanalyse;
- Softwareentwicklung.
SIB ist Mitinitiator der europäischen Bemühungen zur Förderung des offenen Austauschs von SARS-CoV-2-Genomdaten: Le Temps
Die Schweiz richtet eine nationale Infrastruktur zur Verfolgung von COVID-19 und seinen Varianten ein: Le Temps, Tribune de Genève, RTS CQFD, Tages Anzeiger, Heidi.news, Bulletin des médecins suisses
Molekulare Big Data, eine neue Waffe für die Medizin – Experten treffen sich im September in der Schweiz: „Sequenzierung von Krankheitserregern zur Verfolgung von Epidemien“ RTS CQFD
Präzisionsmedizin und die Rolle der Bioinformatik: vier Kurzfilme zum Verständnis: Canal9 (Kurzfilm)
Molekulare Medizin und die CAS in der personalisierten molekularen Onkologie: Bulletin des médecins suisses
- F. Psomopoulos et al.
Toward a unified approach: Considerations for bioinformatic and sequencing activities & data in wastewater surveillance of biologic public health threats.
Open Res Europe 2025, https://open-research-europe.ec.europa.eu/articles/5-267/v1 - M. Ventouratou et al.
The Network of National COVID-19 Data Portals: public health equity through collaboration.
Open Res Europe 2025, https://doi.org/10.12688/openreseurope.20059.1
- Y. Christinat et al.
Reporting of somatic variants in clinical cancer care: recommendations of the Swiss Society of Molecular Pathology.
Virchows Archiv, https://doi.org/10.1007/s00428-024-03951-0 - E. Mutschler et al.
Towards unified reporting of genome sequencing results in clinical microbiology.
PeerJ 12:e17673. https://doi.org/10.7717/peerj.17673 - D. Kraemer et al.
SwissGenVar: A Platform for Clinical-Grade Interpretation of Genetic Variants to Foster Personalized Healthcare in Switzerland.
J. Pers. Med. 2024, 14, 648. https://doi.org/10.3390/jpm14060648 - F. Wegner et al.
How much should we sequence? An analysis of the Swiss SARS-CoV-2 surveillance effort.
Microbiol Spectr. 2024, doi: 10.1128/spectrum.03628-23.
- Aitana Neves et al.
A survey into the contribution of regional/national pathogen data platforms and on the resources needed to develop and maintain them
F1000Research 2023, doi: 10.12688/f1000research.142165.1 - Aitana Neves et al.
FAIR+E pathogen data for surveillance and research: lessons from COVID-19
Frontiers in Public Health, doi: 10.3389/fpubh.2023.1289945 - Aitana Neves et al.
The Swiss Pathogen Surveillance Platform – towards a nation-wide One Health data exchange platform for bacterial, viral and fungal genomics and associated metadata.
Microbial Genomics, doi: 10.1099/mgen.0.001001
- JJC de Vries et al.
Benchmark of Thirteen Bioinformatic Pipelines for Metagenomic Virus Diagnostics Using Datasets from Clinical Samples.
Journal of Clinical Virology : The Official Publication of the Pan American Society for Clinical Virology, doi: 10.1016/j.jcv.2021.104908 - JJC de Vries et al.
Recommendations for the Introduction of Metagenomic Next-Generation Sequencing in Clinical Virology, Part II: Bioinformatic Analysis and Reporting.
Journal of Clinical Virology : The Official Publication of the Pan American Society for Clinical Virology, doi: 10.1016/j.jcv.2021.104812
- G. Greub et al.
Clinical Bioinformatics for Microbial Genomics and Metagenomics: An ESCMID Postgraduate Technical Workshop.
Microbes and Infection, doi: 10.1016/j.micinf.2020.07.008 - D. Dylus et al.
NGS-Based S. aureus Typing and Outbreak Analysis in Clinical Microbiology Laboratories: Lessons Learned From a Swiss-Wide Proficiency Test.
Frontiers 2020, doi: 10.3389/fmicb.2020.591093 - A. David et al.
Annotation and curation of human genomic variations: an ELIXIR Implementation Study.
F1000Research 2020, doi: 10.12688/f1000research.24427.1
- T. Junier et al.
Viral Metagenomics in the Clinical Realm: Lessons Learned from a Swiss-Wide Ring Trial.
Genes 2019, doi: 10.3390/genes10090655
- A. Egli et al.
Improving the quality and workflow of bacterial genome sequencing and analysis: paving the way for a Switzerland-wide molecular epidemiological surveillance platform.
Swiss Med Wkly 2018, doi: 10.4414/smw.2018.14693 - D. Wüthrich et al.
Modern Microbiological Surveillance for Antibiotic Drug Resistance.
Swiss Antibiotic Resistance Report 2018, FOPH publication number: 2018-OEG-87, 93-95 - D. Stekhoven et al.
Swiss Variant Interpretation Platform for Oncology (SVIP-O).
Swiss Med Informatics. 2018, doi: https://doi.org/10.4414/smi.34.00411 - V. Barbié et al.
La santé de demain déjà à la portée des médecins.
Bulletin des Médecins Suisses 2018, doi: https://doi.org/10.4414/bms.2018.06782
- SIB Swiss Institute of Bioinformatics members
The SIB Swiss Institute of Bioinformatics\' resources: focus on curated databases.
Nucleic Acids Res. 2016, doi: 10.1093/nar/gkv1310 - VB Gerritsen et al.
Bioinformatics and personalized medicine: Switzerland is pioneer.
Rev Med Suisse 2016; 12 : 414-6 - JS. Beckmann et al.
Reconciling evidence-based medicine and precision medicine in the era of big data: challenges and opportunities.
Genome Medicine (2016), doi: 10.1186/s13073-016-0388-7
Mitglieder
Unsere Gruppenmitglieder anzeigen hier