Woher stammt das neue Coronavirus (nCoV, siehe Kasten) und wie hat es sich auf den Menschen übertragen? Wie verbreitet es sich und wie entwickelt es sich weiter? Wie können wir Therapien zu seiner Behandlung entwickeln? SIB-Gruppen stellen eine Reihe von Tools und Ressourcen zur Verfügung, die Forschern bei der Beantwortung dieser Fragen helfen können. Diese Tools verknüpfen virale Epidemiologie und Genomik mit Proteinsequenzen, Varianten, Funktionen und dreidimensionalen Strukturen. Im Folgenden erfahren Sie mehr über einige dieser Tools.
Coronavirus: einige biologische Fakten
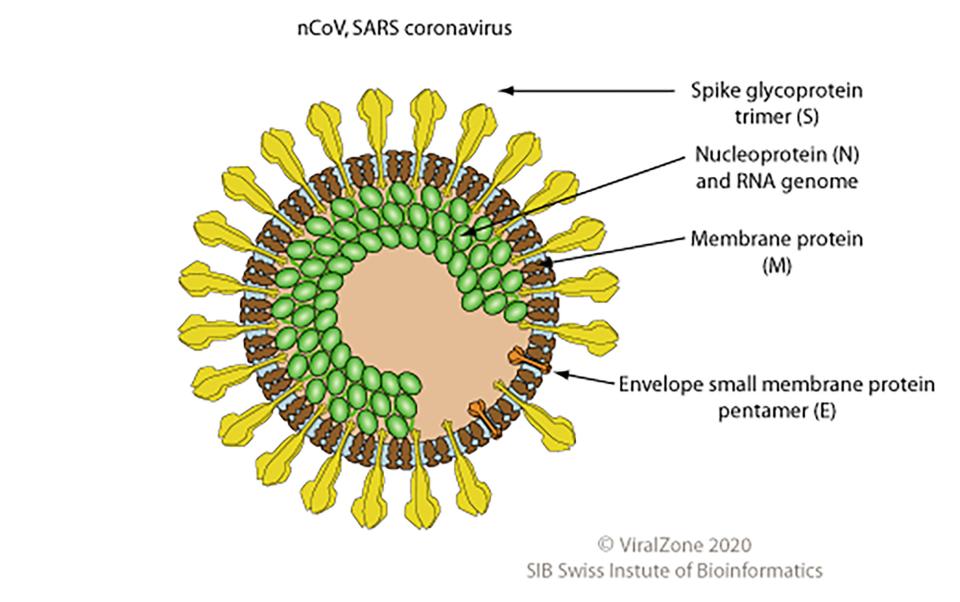
Das Coronavirus 2019–20, das erstmals in Wuhan, der Hauptstadt der chinesischen Provinz Hubei, identifiziert wurde, ist ein umhülltes einzelsträngiges RNA-Virus. Der Name „Coronavirus“ leitet sich vom lateinischen Wort „corona“ (Krone) ab und bezieht sich sehr treffend auf die Form der Proteine um das Virion (siehe Abbildung). Coronaviren kommen bei vielen Wirbeltierarten vor und verursachen Atemwegserkrankungen wie die Erkältung oder SARS. Das neuere Coronavirus nCoV stammt aus einem noch unbekannten Tierreservoir und kann von Mensch zu Mensch übertragen werden.
Das Coronavirus kennenlernen: von der Genomik bis zur Epidemiologie…
Die erste Genomsequenz des Coronavirus wurde am 10. Januar 2020 veröffentlicht. Seitdem werden die nCoV-Genome, sobald sie öffentlich zugänglich sind, in die von der SIB-Gruppe um Richard Neher (Universität Basel) mitentwickelte RessourceNextStrain aufgenommen. Damit können klinische Forscher nahezu in Echtzeit verfolgen, wie sich das Coronavirus-Genom entwickelt – Informationen, die für die Bekämpfung der Ausbreitung der Krankheit von entscheidender Bedeutung sind.
…zum Wissen über virale Proteine, ihre Funktionen und Strukturen…
Das Coronavirus-Genom kodiert 13 (oder möglicherweise 14) Proteine. Diese viralen Proteine steuern die Transkription, Replikation und Ausbreitung des Virus in der Wirtspopulation.
Das Wissen über virale Proteinsequenzen und deren Funktionsweise ist nun als Vorabversion der Coronavirus-Einträge der universellen Proteinkenntnisdatenbank UniProtverfügbar – einer internationalen Zusammenarbeit zwischen der Swiss-Prot-Gruppe der SIB unter der Leitung von Alan Bridge, EMBL-EBI, und PIR.
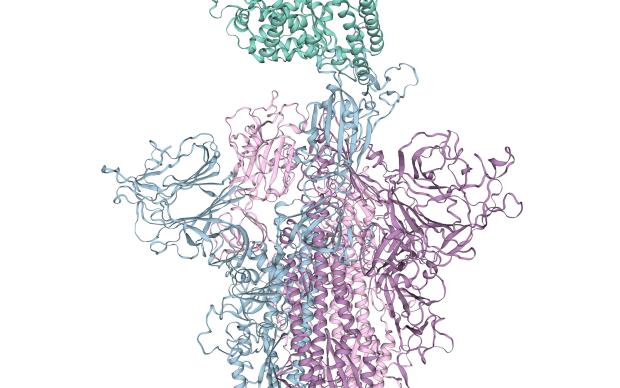
In Kombination mit Daten zur Evolution des Virusgenoms und zur Epidemiologie werden die Kenntnisse über virale Proteine für klinische Forscher bei der Entwicklung neuer Therapien von entscheidender Bedeutung sein. SWISS-MODEL, entwickelt von der SIB-Gruppe um Torsten Schwede an der Universität Basel, kann beispielsweise dreidimensionale Modelle dieser viralen Proteine erstellen (siehe eigene Seite) – Modelle, die weitere Aufschlüsse über ihre Evolution und funktionellen Eigenschaften geben (wie in diesen beiden aktuellen Beispielen hier und hier) – und möglicherweise auch über ihre Schwachstellen im Hinblick auf die Entwicklung von Medikamenten.
Eine neue Seite zu nCoV auf ViralZone bietet weitere biologische Einblicke, darunter einen detaillierten Vergleich mit dem Genom des SARS-Virus sowie Querverweise zu den oben genannten ergänzenden Ressourcen. „Wir heben die spezifischen Unterschiede zwischen nCoV und SARS hervor, insbesondere in der Genzusammensetzung, die näher an Fledermausviren zu liegen scheint und möglicherweise die Unterschiede in der Pathogenität und Übertragung erklärt“, erklärt Philippe Le Mercier, der die Entwicklung von ViralZone bei der Swiss-Prot-Gruppe der SIB leitet.