Wissenschaftler des SIB und der Universität Basel sowie von Chelonia Applied Sciences (Basel) sind Teil einer europäischen Initiative, die nach Wirkstoffen gegen COVID-19 sucht – und diesen Prozess in Zukunft beschleunigen will. Sie sind Teil von Exscalate4CoV (E4C), einem öffentlich-privaten Konsortium aus 18 führenden EU-Organisationen – darunter die europäische Supercomputing-Plattform EXSCALATE – aus sieben Ländern, das von der Europäischen Kommission im Rahmen von Horizon 2020 mit drei Millionen Euro gefördert wird. Die Wissenschaftler werden die SIB-Ressource SWISS-MODEL nutzen, um 3D-Modelle von Proteinen zu erstellen, die noch nicht experimentell aufgeklärt sind, und so das virtuelle Screening nach potenziellen Wirkstoffen beschleunigen.
Beschleunigung der Suche nach Medikamenten gegen COVID-19
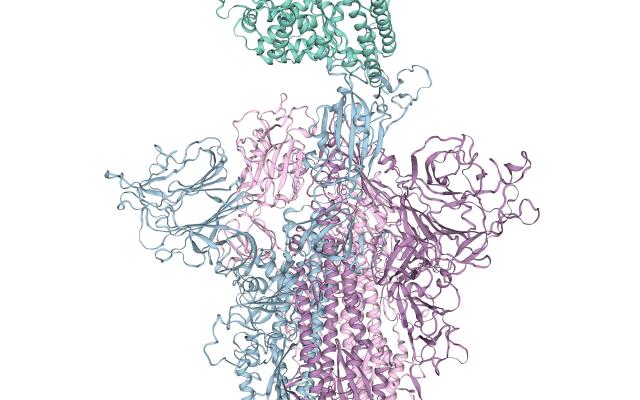
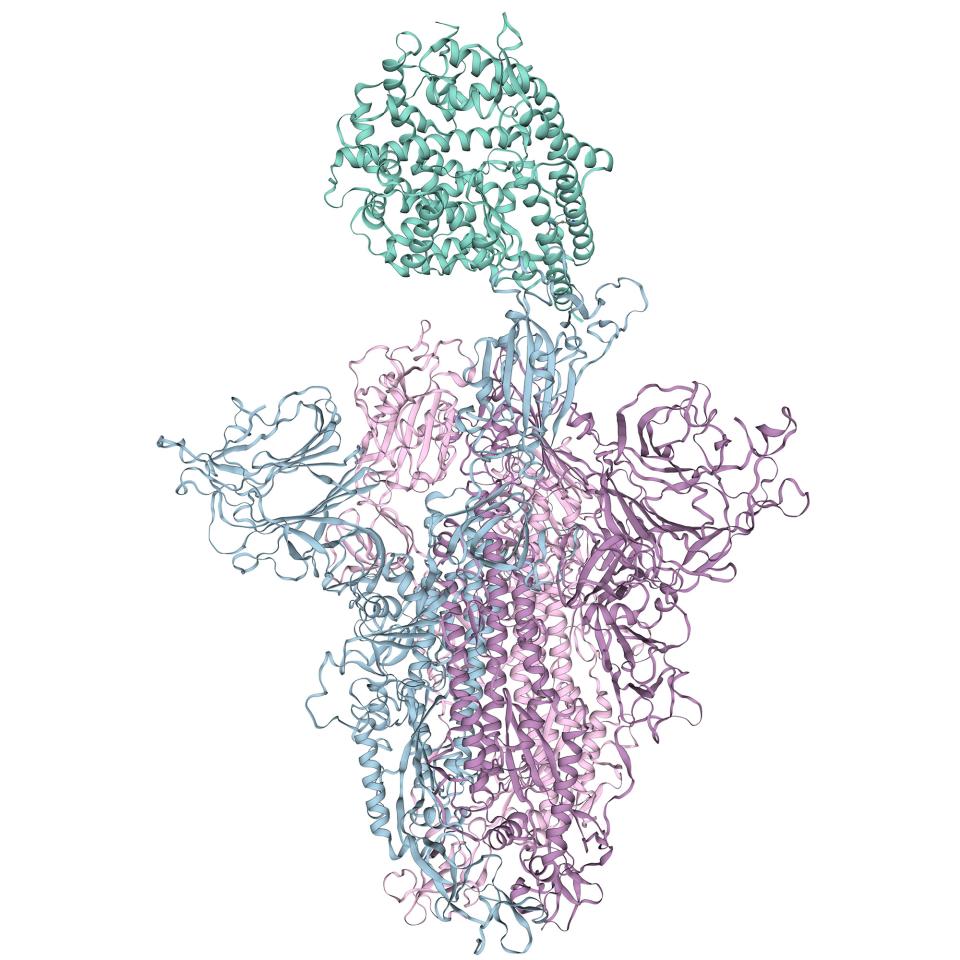
Als Teil des E4C-Konsortiums ist die SIB Computational Structural Biology (CSB) Group am Biozentrum (Universität Basel) für die Erstellung von in silico vorhergesagten dreidimensionalen Modellen von COVID-19-Proteinen zuständig . „Da es sich um ein relativ neues Virus handelt, haben Wissenschaftler erst vor kurzem damit begonnen, die 3D-Strukturen der viralen Proteine experimentell zu charakterisieren. Mit Hilfe von Computermodellen lassen sich Strukturen anhand von Informationen aus verwandten Proteinen, beispielsweise aus SARS-CoV, modellieren, wodurch der Prozess der Wirkstoffforschung beschleunigt wird“, erklärt Gerardo Tauriello, Leiter des Entwicklungsteams bei CSB. Dieser Ansatz wird als Homologiemodellierung bezeichnet, und eines der führenden Werkzeuge dafür wurde vom Team entwickelt: der SWISS-MODEL-Server, eine frei zugängliche Ressource der SIB. Sowohl experimentelle Strukturen als auch Homologiemodelle werden von den anderen Teilnehmern von E4C verwendet, um kleine Moleküle zu identifizieren, die potenziell gegen das Coronavirus wirken könnten. Diese Moleküle werden dann in In-vitro-Experimenten, Tiermodellen und – bei Erfolg – in klinischen Studien weiter getestet .
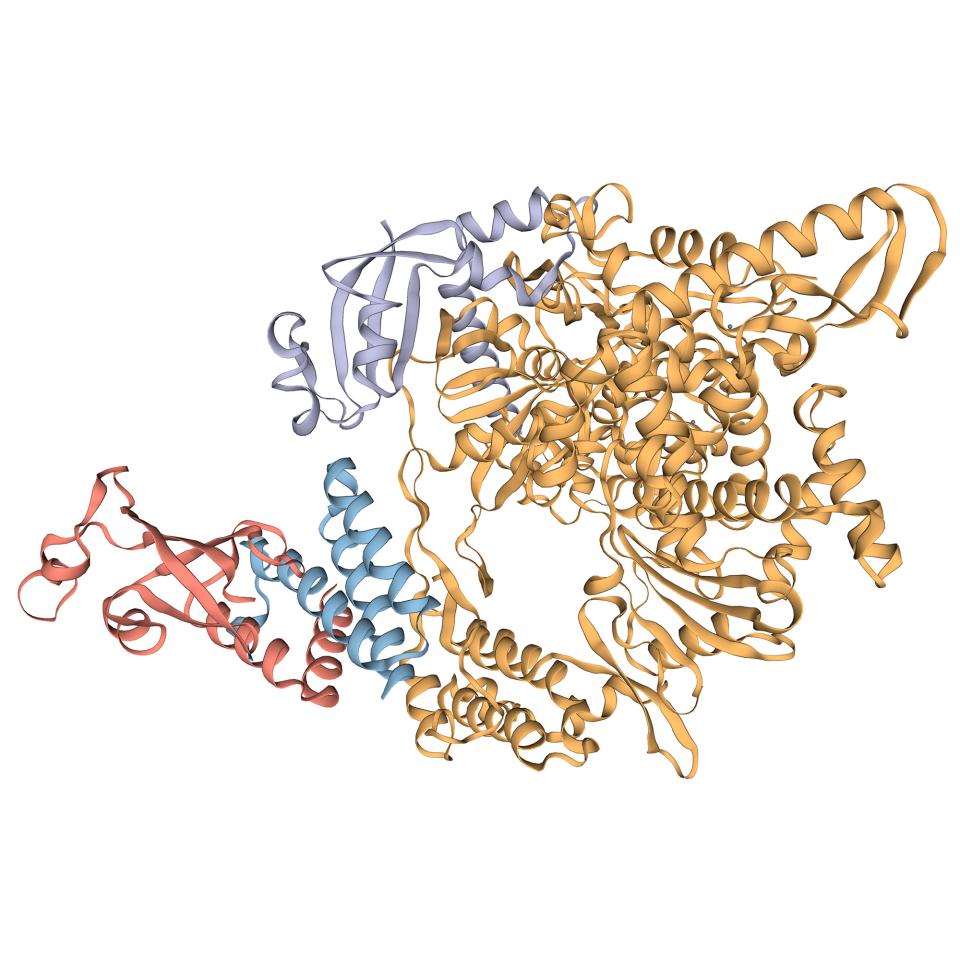
Proteine in 3D untersuchen, um die Entwicklung neuer Medikamente zu unterstützen
Viren sind zwar auf die Maschinerie der Wirtszellen angewiesen, um ihr Genom zu exprimieren, aber sie sind ebenso wie lebende Organismen von Proteinen abhängig, die in ihrem Genom kodiert sind. Jedes Protein hat eine bestimmte dreidimensionale Struktur, die eng mit seiner Funktion verbunden ist. Beispielsweise werden vom Virus verschiedene Transmembranproteine verwendet, um sich an Wirtszellen zu binden. Wirkstoffmoleküle sind so konzipiert, dass sie an relevante Teile eines Proteins binden, um dessen Funktion zu modulieren. Eine dreidimensionale Darstellung der Proteinstruktur auf atomarer Ebene ist daher ein wichtiger Aspekt bei der rationalen Wirkstoffforschung.
Jenseits von COVID-19: Schnellere Entwicklung von Medikamenten für künftige Pandemien
E4C will die Erkenntnisse und Entwicklungen aus der aktuellen Pandemie nutzen, um die Entdeckung und Identifizierung von Medikamenten im Falle künftiger Pandemien zu beschleunigen – insbesondere wenn der Erreger wissenschaftlich unbekannt ist und noch keine Behandlungsmöglichkeiten existieren. „Heute erfordert die Verarbeitung eines neuartigen Virusgenoms zeitaufwändige manuelle Arbeit durch Modellierungsexperten, was die Reaktionszeit einschränkt. In Zukunft wird jeder Forscher, sobald ein neues Virus sequenziert ist, mit SWISS-MODEL innerhalb von Minuten Modelle erstellen können, die dann auf der EXSCALATE-Plattform zur Verwendung und Interpretation bereitstehen“, erklärt Tauriello. Im Rahmen von E4C wird die SIB CSB-Gruppe einen automatisierten und skalierbaren Workflow entwickeln und benchmarken, der die Erstellung von Proteinmodellen durch Homologie direkt aus dem Genom eines beliebigen Virus ermöglicht.
Kopplung von führenden Life-Science-Forschungslabors mit leistungsstarken Supercomputern
E4C verbindet die europäische Supercomputing-Plattform EXSCALATE (EXaSCale smArt pLatform Against paThogEns), die dank einer Verarbeitungskapazität von mehr als 3 Millionen Molekülen pro Sekunde auf eine Bibliothek mit 500 Milliarden Wirkstoffkandidaten zurückgreifen kann, mit führenden Forschungslabors im Bereich der Biowissenschaften. Chelonia Applied Science mit Sitz in Basel hat das E4C-Konsortium konzipiert und ist für dessen Verbreitung und Verwertung zuständig. Silvano Coletti, Chief Executive Officer von Chelonia, sagt: „Die Situation, mit der die Welt aufgrund der COVID-19-Pandemie konfrontiert ist, ist in unserer Lebenszeit beispiellos und betrifft uns alle beruflich und persönlich. Exscalate4CoV ist ein ebenso beispielloses Rechenprojekt, das darauf abzielt, die Identifizierung von COVID-19-Medikamenten heute zu beschleunigen und morgen zur Referenzplattform für zukünftige Pandemien zu werden – einschließlich des Entdeckungs- und Entwicklungsprozesses in der pharmazeutischen Forschung.“
Neben den Schweizer Mitgliedern umfasst das E4C-Konsortium unter der Koordination des italienischen Pharmaunternehmens Dompé 16 Institutionen* aus sechs Ländern. Weitere Informationen finden Sie auf der Website von EXSCALATE4CoV.
*Dompé Farmaceutici SPA, Politecnico di Milano (Fachbereich für Elektronik, Information und Bioingenieurwesen), Consorzio Interuniversitario CINECA (Supercomputing Innovation and Applications), Università degli Studi di Milano (Fachbereich Pharmazeutische Wissenschaften), Katholieke Universiteit Leuven, International Institute Of Molecular And Cell Biology In Warsaw (LIMCB), Elettra Sincrotrone Trieste, Fraunhofer-Institut für Molekularbiologie und Angewandte Ökologie, Bsc Supercomputing Centre, Forschungszentrum Jülich, Universität Federico II di Napoli, Universität Cagliari, KTH Royal Institute of Technology (Fachbereich Angewandte Physik), Associazione BigData, Istituto Nazionale di Fisica Nucleare (INFN) und Istituto nazionale per le malattie infettive Lazzaro Spallanzani.