Künstliche Intelligenz hat ihre ersten Nobelpreise gewonnen – darunter für bahnbrechende Werkzeuge in den Lebenswissenschaften – und generative KI wird immer leistungsfähiger und verbreiteter. Das SIB fördert, ermöglicht und nutzt diese und andere transformative Fortschritte in der KI.
SIB-Fachwissen und -Daten entscheidend für Nobelpreis-gekröntes KI-Modell
AlphaFold wurde für seine Fähigkeit, 3D-Proteinstrukturen anhand ihrer Aminosäuresequenz vorherzusagen, mit dem Nobelpreis für Chemie 2024 ausgezeichnet. Das Modell basiert auf jahrzehntelanger Bioinformatik-Expertise – darunter drei offene Ressourcen und Initiativen, die von Wissenschaftlern des SIB entwickelt und mitentwickelt wurden:
- Das KI-Modell lernte, Beziehungen zwischen Aminosäuresequenzen und 3D-Strukturen zu identifizieren, indem es Hunderte Millionen hochwertiger Proteinsequenzen in UniProt analysierte. Expertenkommentare zur Proteinstruktur halfen den Entwicklern von AlphaFold außerdem, die Modellleistung zu verstehen und zu optimieren.
- Seine beeindruckende Genauigkeit wurde erstmals von CASP demonstriert – einem globalen Wettbewerb, der alle zwei Jahre stattfindet und die Genauigkeit von Berechnungsmethoden anhand experimentell ermittelter, unveröffentlichter und sehr anspruchsvoller Proteinstrukturen testet. Torsten Schwede, Gruppenleiter am SIB, ist seit 2011 Mitglied des CASP-Organisationskomitees.
- Die Anwendbarkeit von AlphaFold auf alle menschlichen Proteine wurde anschließend anhand von Daten aus CAMEO bestätigt, das denselben Genauigkeitstest wie CASP durchführt, jedoch mit einem größeren Satz von Proteinen, die wöchentlich veröffentlicht werden.
Sehen Sie, wie SIB-Wissenschaftler das Proteinuniversum anhand von AlphaFold-Daten kartiert haben.
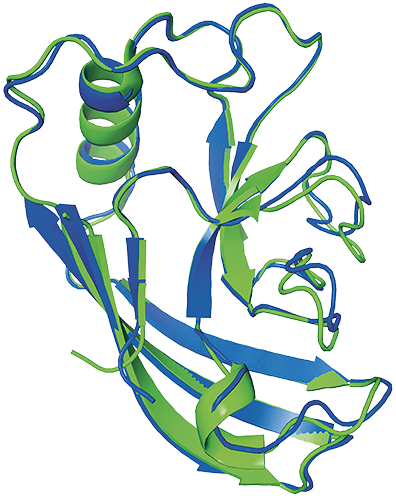
Von AlphaFold vorhergesagte Proteinstruktur (blau) und experimentell ermittelte Proteinstruktur (grün)
Bereitstellung von Goldstandard-Trainingsdaten durch kuratierte Datenbanken
Die kuratierten Datenbanken des SIB liefern äußerst zuverlässige Daten und Erkenntnisse, anhand derer KI-Modelle lernen können, Muster zu erkennen und relevante Vorhersagen zu treffen. Eine dieser Datenbanken, UniProt, war für das Training von AlphaFold von entscheidender Bedeutung (siehe Kasten). Viele weitere Datenbanken stehen für KI-Anwendungen zur Verfügung und werden dort genutzt, um komplexe Herausforderungen in den Lebenswissenschaften zu bewältigen.
SIB-Fachwissen und -Daten entscheidend für Nobelpreis-gekröntes KI-Modell
AlphaFold wurde für seine Fähigkeit, 3D-Proteinstrukturen anhand ihrer Aminosäuresequenz vorherzusagen, mit dem Nobelpreis für Chemie 2024 ausgezeichnet. Das Modell basiert auf jahrzehntelanger Bioinformatik-Expertise – darunter drei offene Ressourcen und Initiativen, die von Wissenschaftlern des SIB entwickelt und mitentwickelt wurden:
- Das KI-Modell lernte, Beziehungen zwischen Aminosäuresequenzen und 3D-Strukturen zu identifizieren, indem es Hunderte Millionen hochwertiger Proteinsequenzen in UniProt analysierte. Expertenkommentare zur Proteinstruktur halfen den Entwicklern von AlphaFold außerdem, die Modellleistung zu verstehen und zu optimieren.
- Seine beeindruckende Genauigkeit wurde erstmals von CASP demonstriert – einem globalen Wettbewerb, der alle zwei Jahre stattfindet und die Genauigkeit von Berechnungsmethoden anhand experimentell ermittelter, unveröffentlichter und sehr anspruchsvoller Proteinstrukturen testet. Torsten Schwede, Gruppenleiter am SIB, ist seit 2011 Mitglied des CASP-Organisationskomitees.
- Die Anwendbarkeit von AlphaFold auf alle menschlichen Proteine wurde anschließend anhand von Daten aus CAMEO bestätigt, das denselben Genauigkeitstest wie CASP durchführt, jedoch mit einem größeren Satz von Proteinen, die wöchentlich veröffentlicht werden.
Sehen Sie, wie SIB-Wissenschaftler das Proteinuniversum anhand von AlphaFold-Daten kartiert haben.

Von AlphaFold vorhergesagte Proteinstruktur (blau) und experimentell ermittelte Proteinstruktur (grün)
- Umwandlung von Daten in KI-fähiges Wissen
Unsere Datenbanken sind offen, FAIR und maschinenlesbar, was durch harmonisierte Daten, reichhaltige Metadaten und strenge Qualitätskontrollen erreicht wird.
Fachmännisch kuratierte Datenbanken enthalten zusätzlich relevante und kontinuierlich aktualisierte Informationen aus der wissenschaftlichen Literatur und anderen Quellen. Unsere Biokuratoren versehen beispielsweise Proteinsequenzen in UniProt mit Anmerkungen zu Struktur, Funktion und weiteren Eigenschaften der Proteine.
Diese explizite Kodierung komplexer Informationen in maschinenlesbaren Formaten ist für Fortschritte der KI im wissenschaftlichen Bereich unerlässlich.- Eine Fülle frei verfügbarer Trainingsdaten
SIB entwickelt gemeinsam offene, kuratierte Datenbanken, die Biomoleküle und Prozesse aus dem gesamten Stammbaum des Lebens umfassen.
Beispiele hierfür sind Genexpression (Bgee), Zelllinien (Cellosaurus), Kohlenhydrate (Glyco@Expasy), Reaktionen (Rhea), Proteininteraktionen (STRING), Lipide (SwissLipids), Pathogengenome (SPSP), Orthologe (SwissOrthology), Proteinsequenzen und -strukturen (UniProt, Swiss-Model) sowie Genfunktionen (Gene Ontology).
- Eine Fülle frei verfügbarer Trainingsdaten
- Ermöglichung von Schulungen zu sensiblen Daten
Wir entwickeln auch ethische, sichere Methoden für KI-Analysen von menschlichen Daten, die nicht offen geteilt werden können. Die föderierte Analyse ermöglicht beispielsweise den Zugriff auf relevante Daten in einer sicheren Umgebung, ohne dass personenbezogene Daten weitergegeben werden.
Wissenschaftler der SIB verfolgen diesen Ansatz in mehreren Initiativen, darunter die internationale Initiative iCARE4CVD zur personalisierten Prävention und Behandlung von Herz-Kreislauf-Erkrankungen (mehr dazu) und BioMedIT, die vertrauenswürdige Forschungsumgebung des Swiss Personalized Health Network (SPHN; mehr dazu).
Umsetzbare Erkenntnisse durch Wissensrepräsentation ermöglichen
Eine einzelne Datenbank ist weitaus leistungsfähiger, wenn das darin enthaltene Wissen mit anderen Datenbanken verknüpft ist. Wissenschaftler der SIB überbrücken Datensilos zwischen verschiedenen Fachgebieten und Ländern, indem sie Systeme zur formalen Darstellung von Wissen entwickeln und implementieren – beispielsweise standardisierte Vokabulare zur Darstellung biologischer Einheiten und ihrer Beziehungen (z. B. Gene, Proteine, Metaboliten, Arten, Krankheiten).
Dies ermöglicht es KI und Forschern, Daten aus verschiedenen Quellen effektiv abzurufen und zu verarbeiten, Verbindungen zwischen diesen Daten aufzudecken und so einen ganzheitlichen, integrierten Überblick über komplexe biologische Systeme zu gewinnen.
Beispiele hierfür sind:
- Entwicklung einer KI-basierten Suche über die gesamte Sammlung europäischer Forschungsergebnisse hinweg durch das Projekt „European Open Science Cloud (EOSC) Data Commons” (weitere Informationen);
- die Ermöglichung einer KI-gesteuerten Präzisionsonkologie durch die Umwandlung unstrukturierter PDF-Richtlinien für die Krebsbehandlung in strukturierte, interoperable Behandlungsverfahren und die Entwicklung eines KI-Tools zur Analyse der Verfahren sowie historischer klinischer Daten, um die beste Behandlung für einzelne Patienten vorherzusagen, im Rahmen des AI Tumor Board-Projekts mit Schweizer Krankenhäusern;
- die Förderung der KI-gestützten Abfrage von kuratiertem Pflanzenwissen durch die Verknüpfung chemischer Daten über Pflanzen mit Informationen wie Arteninteraktionen und Merkmalen im Rahmen der Digital Botanical Gardens Initiative.
Weitere Informationen zur Wissensrepräsentation finden Sie unter SIB.
Sicherstellung vertrauenswürdiger Ergebnisse durch Benchmarking
Wie können KI-Entwickler – und die Nutzer ihrer Modelle – Vertrauen in KI-Prognosen haben? Und wie können Forscher wissen, welches KI-Tool für eine bestimmte Aufgabe am besten geeignet ist? Die Benchmarking-Expertise von SIB hat diese Fragen für AlphaFold beantwortet (siehe Kasten oben) und ermöglicht eine strenge Bewertung und Feinabstimmung vieler anderer KI- und computergestützter Analysen.
Unsere Wissenschaftler
- stellen Referenzdatensätze nach Goldstandard bereit, die als standardisierte Eingaben für den Vergleich von KI-Tools, als Zielergebnisse, anhand derer Vorhersagen bewertet werden können, und als Ressourcen für die Feinabstimmung von Modellen für bestimmte Bereiche oder die Bewertung der Datenqualität vor der Analyse dienen;
- entwickeln Benchmarking-Softwaretools, um die KI-Leistung anhand von Referenzdaten oder anderen Modellen zu bewerten, Verbesserungsmöglichkeiten aufzuzeigen und Forschern bei der Auswahl des für ihre Bedürfnisse am besten geeigneten Modells zu helfen.
Beispiele hierfür sind Tools und Datensätze zur Bewertung von Vorhersagen zur Proteinfaltung und Proteininteraktion (CAMEO und CASP; siehe Kasten oben), zur Proteomqualität (SwissOrthology) und zur Genom- und Metagenomqualität (BUSCO und LEMMI); einen Datensatz zur Feinabstimmung generativer KI-Modelle (siehe unten) für die spezialisierte Datenkuratierung (EnzChemRED); und ein System, das eine reproduzierbare, neutrale Benchmarking von Tools für die computergestützte Biologie nach den FAIR-Prinzipien (Findable, Accessible, Interoperable and Reusable) gewährleistet (Omnibenchmark).
Von generativer KI und LLMs
Generative KI umfasst Systeme, die in der Lage sind, neue Inhalte zu erstellen, von Texten und Bildern bis hin zu Videos, Musik und vielem mehr. Große Sprachmodelle (LLMs), eine wichtige Art der generativen KI, werden anhand umfangreicher Textdaten, darunter genetische Sequenzen oder Informatikcode, trainiert, um neue Inhalte zusammenzufassen, zu generieren und vorherzusagen. Modelle wie ChatGPT und BioBERT sind Beispiele dafür, wobei ChatGPT sich durch die Generierung von Text für Chatbots und kreatives Schreiben auszeichnet, während BioBERT sich auf biomedizinische Texte konzentriert (d. h. darauf vorab trainiert wurde). LLMs verwenden Deep-Learning-Techniken, insbesondere Transformer, um Sprachmuster aus riesigen Datensätzen zu analysieren und zu verstehen und das nächste „Wort” oder die nächste Wortfolge basierend auf dem Kontext vorherzusagen.
Beschleunigung von Entdeckungen in den Lebenswissenschaften durch generative KI
ChatGPT und eine Vielzahl anderer generativer KI-Modelle verändern nicht nur unseren Alltag, sondern auch die Wissenschaft. SIB begrüßt diese Revolution in allen Bereichen, von klinischen Anwendungen bis hin zur Generierung biologischer Erkenntnisse.
Von generativer KI und LLMs
Generative KI umfasst Systeme, die in der Lage sind, neue Inhalte zu erstellen, von Texten und Bildern bis hin zu Videos, Musik und vielem mehr. Große Sprachmodelle (LLMs), eine wichtige Art der generativen KI, werden anhand umfangreicher Textdaten, darunter genetische Sequenzen oder Informatikcode, trainiert, um neue Inhalte zusammenzufassen, zu generieren und vorherzusagen. Modelle wie ChatGPT und BioBERT sind Beispiele dafür, wobei ChatGPT sich durch die Generierung von Text für Chatbots und kreatives Schreiben auszeichnet, während BioBERT sich auf biomedizinische Texte konzentriert (d. h. darauf vorab trainiert wurde). LLMs verwenden Deep-Learning-Techniken, insbesondere Transformer, um Sprachmuster aus riesigen Datensätzen zu analysieren und zu verstehen und das nächste „Wort” oder die nächste Wortfolge basierend auf dem Kontext vorherzusagen.
Beispiele hierfür sind:
- Beschleunigung biologischer und bioinformatischer Entdeckungen mit ExpasyGPT – einem maßgeschneiderten generativen KI-Tool, das in Expasy, das Schweizer Bioinformatik-Ressourcenportal, integriert ist und es Forschern ermöglicht, Informationen aus SIB-Datenbanken schneller und einfacher als je zuvor abzurufen und zusammenzustellen (mehr dazu);
- die schnelle Generierung maßgeschneiderter Antikörper zur Bekämpfung von Krankheiten durch AntibodyGPT, das den traditionell langsamen Prozess der Entdeckung monoklonaler Antikörper beschleunigt, indem es Antikörperstrukturen mit den gewünschten Eigenschaften vorhersagt;
- Testen der Fähigkeit von ChatGPT, medizinische Fragen in der Strahlentherapie zu beantworten (siehe Veröffentlichung);
- Entschlüsselung der verborgenen Rolle von RNA bei Krebserkrankungen durch Fachwissen im Bereich der natürlichen Sprachverarbeitung;
- Verständnis der Häutung von Insekten durch die Integration von Artennamendaten mit Sequenzdaten aus verschiedenen öffentlichen Datenbanken unter Verwendung generativer KI-Methoden in die Ressource MoultDB, die als Referenz für diesen Bereich dient.
Sehen Sie, wie SIB-Experten Herausforderungen mit generativer KI angehen.
Generative KI und Biokuration: ein positiver Kreislauf
Das Zusammenspiel zwischen den Möglichkeiten der KI, insbesondere der LLM, und der Bedeutung des menschlichen Fachwissens lässt sich gut am Beispiel der Biokuration veranschaulichen, bei der das SIB anerkanntermaßen führend ist. Biokuration ist die Kunst der fachkundigen Extraktion von Wissen aus der biologischen und biomedizinischen Literatur, um eine genaue, zuverlässige und aktuelle Enzyklopädie zu erstellen, die der Wissenschaft im Allgemeinen dient.
Ausführliche Berichte über vertrauenswürdige KI
- Die KI-Revolution geht weiter – SIB Profile 2025
- Generative KI: neue Horizonte – SIB Profile 2024
- Rund um maschinelles Lernen – SIB Profile 2021