D'où vient le nouveau coronavirus (nCoV, voir encadré) et comment s'est-il transmis à l'homme ? Comment se propage-t-il et évolue-t-il ? Comment développer des traitements pour le soigner ? Les groupes SIB fournissent une gamme d'outils et de ressources qui peuvent aider les chercheurs à répondre à ces questions. Ces outils relient l'épidémiologie virale et la génomique aux séquences protéiques, aux variants, aux fonctions et aux structures tridimensionnelles. Découvrez ci-dessous certains de ces outils.
Coronavirus : quelques faits biologiques
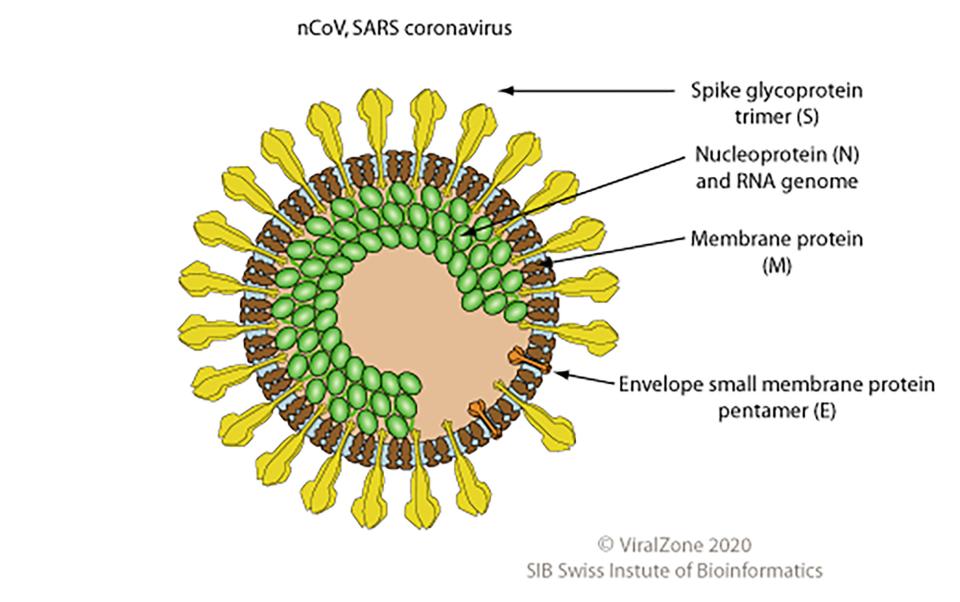
Le coronavirus 2019-2020, identifié pour la première fois à Wuhan, capitale de la province chinoise du Hubei, est un virus à ARN simple brin enveloppé. Le nom « coronavirus » vient du latin corona (couronne) et fait référence à la forme des protéines qui entourent le virion (voir image). Les coronavirus sont présents chez de nombreuses espèces vertébrées et provoquent des maladies respiratoires, telles que le rhume ou le SRAS. Le coronavirus nCoV, plus récent, est apparu à partir d'un réservoir animal encore inconnu et peut se transmettre d'une personne à l'autre.
Comprendre le coronavirus : de la génomique à l'épidémiologie..
La première séquence du génome du coronavirus a été publiée le 10 janvier 2020. Depuis lors, la ressource NextStrain, développée conjointement par le groupe SIB de Richard Neher (Université de Bâle), intègre les génomes du nCoV dès qu'ils sont rendus publics et permet aux chercheurs cliniques de suivre en temps quasi réel l'évolution du génome du coronavirus, une information qui sera cruciale dans la lutte contre la propagation de la maladie.
…à la connaissance des protéines virales, de leurs fonctions et de leurs structures…
Le génome du coronavirus code 13 (voire 14) protéines. Ces protéines virales contrôlent la transcription, la réplication et la propagation du virus dans la population hôte.
Les séquences des protéines virales et leur fonctionnement sont désormais disponibles dans une version préliminaire des entrées consacrées au coronavirus de la base de données universelle sur les protéines UniProt, fruit d'une collaboration internationale entre le groupe Swiss-Prot du SIB, dirigé par Alan Bridge, l'EMBL-EBI et le PIR.
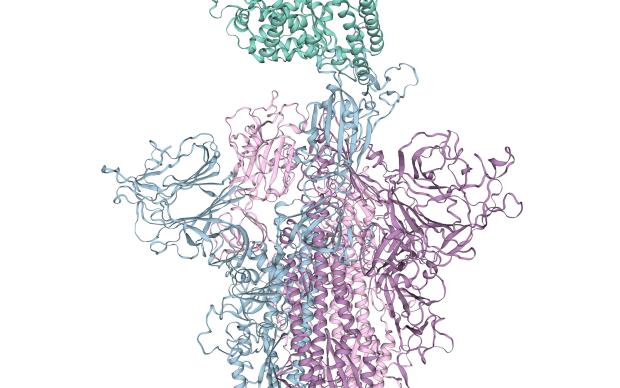
Combinées aux données sur l'évolution du génome viral et l'épidémiologie, les connaissances sur les protéines virales seront cruciales pour aider les chercheurs cliniques dans leurs efforts pour concevoir de nouvelles thérapies. SWISS-MODEL, développé par le groupe SIB de Torsten Schwede à l'Université de Bâle, peut par exemple générer des modèles tridimensionnels de ces protéines virales (voir page dédiée)– des modèles qui apportent un éclairage supplémentaire sur leur évolution et leurs propriétés fonctionnelles (comme dans ces deux exemples récents ici et ici)– et, potentiellement, sur leurs faiblesses, dans le contexte du développement de médicaments.
Une nouvelle page consacrée au nCoV sur ViralZone fournit des informations biologiques supplémentaires, notamment une comparaison détaillée avec le génome du virus du SRAS, ainsi que des liens vers les ressources complémentaires mentionnées ci-dessus. « Nous mettons en évidence les différences spécifiques entre le nCoV et le SARS, en particulier dans sa composition génétique qui semble plus proche des virus de chauves-souris et pourrait expliquer la différence de pathogénicité et de transmission », explique Philippe Le Mercier, qui dirige le développement de ViralZone au sein du groupe Swiss-Prot du SIB.