Da dove ha avuto origine il nuovo coronavirus (nCoV, vedi riquadro) e come si è trasmesso agli esseri umani? Come si sta diffondendo ed evolvendo? Come possiamo sviluppare terapie per curarlo? Il SIB fornisce una serie di strumenti e risorse che possono aiutare i ricercatori a rispondere a queste domande. Questi strumenti collegano l'epidemiologia virale e la genomica alle sequenze proteiche, alle varianti, alle funzioni e alle strutture tridimensionali. Scopri di più su alcuni di questi strumenti qui sotto.
Coronavirus: alcuni fatti biologici
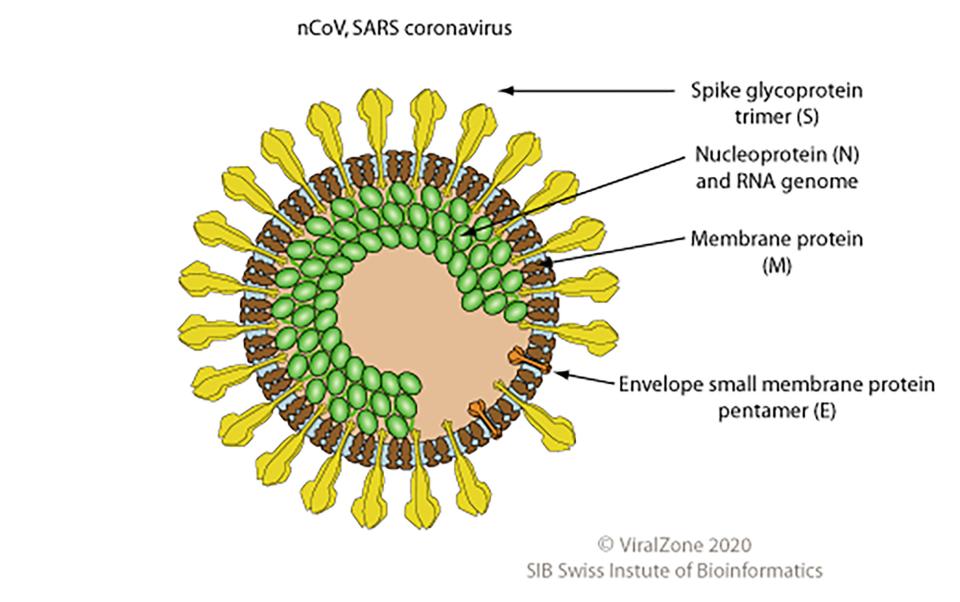
Il coronavirus 2019-20, identificato per la prima volta a Wuhan, capitale della provincia cinese di Hubei, è un virus a RNA a singolo filamento con involucro. Il nome "Coronavirus" deriva dal latino corona (cioè corona) e si riferisce in modo molto appropriato alla forma delle proteine che circondano il virione (vedi immagine). I coronavirus sono presenti in molte specie di vertebrati e causano malattie respiratorie, come il comune raffreddore o la SARS. Il più recente coronavirus nCoV è emerso da un serbatoio animale ancora sconosciuto e può essere trasmesso da uomo a uomo.
Conoscere il coronavirus: dalla genomica all'epidemiologia..
La prima sequenza del genoma del coronavirus è stata pubblicata il 10 gennaio 2020. Da allora, la risorsaNextstrain, sviluppata in collaborazione con il gruppo del SIB di Richard Neher (Università di Basilea), incorpora i genomi nCoV non appena vengono resi pubblici e consente ai ricercatori clinici di seguire in tempo quasi reale l'evoluzione del genoma del coronavirus, un'informazione che sarà fondamentale nella lotta contro la diffusione della malattia.
...alla conoscenza delle proteine virali, delle loro funzioni e strutture..
Il genoma del coronavirus codifica 13 (o forse 14) proteine. Queste proteine virali controllano la trascrizione, la replicazione e la diffusione del virus nella popolazione ospite.
Le conoscenze relative alle sequenze delle proteine virali e al loro funzionamento sono ora disponibili in anteprima nelle voci dedicate al coronavirus della banca dati universale sulle proteineUniProt, frutto di una collaborazione internazionale tra il gruppo Swiss-Prot del SIB, guidato da Alan Bridge, l'EMBL-EBI e il PIR.
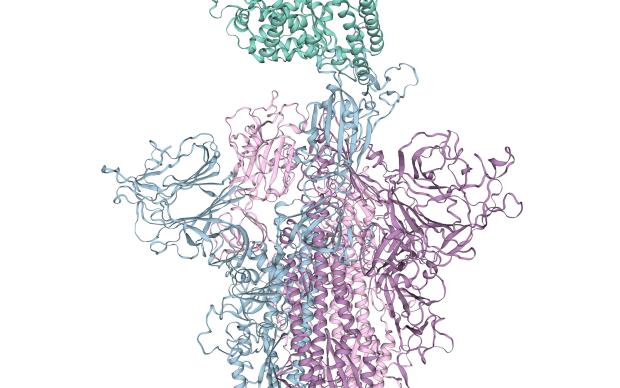
In combinazione con i dati sull'evoluzione del genoma virale e sull'epidemiologia, la conoscenza delle proteine virali sarà fondamentale per aiutare i ricercatori clinici nei loro sforzi per progettare nuove terapie. SWISS-MODEL, sviluppato dal gruppo il SIB di Torsten Schwede dell'Università di Basilea, può ad esempio generare modelli tridimensionali di tali proteine virali (vedi pagina dedicata)- modelli che gettano ulteriore luce sulla loro evoluzione e sulle loro proprietà funzionali (come in questi due recenti esempi qui e qui)- e, potenzialmente, sui loro punti deboli, nel contesto dello sviluppo di farmaci.
Una nuova pagina dedicata al nCoV su ViralZone fornisce ulteriori approfondimenti biologici, tra cui un confronto dettagliato con il genoma del virus della SARS e collegamenti incrociati alle risorse complementari sopra menzionate. "Stiamo evidenziando le differenze specifiche tra nCoV e SARS, in particolare nella composizione genetica che sembra più vicina ai virus dei pipistrelli e potrebbe spiegare la differenza in termini di patogenicità e trasmissione", spiega Philippe Le Mercier, responsabile dello sviluppo di ViralZone presso il gruppo Swiss-Prot di al SIB.