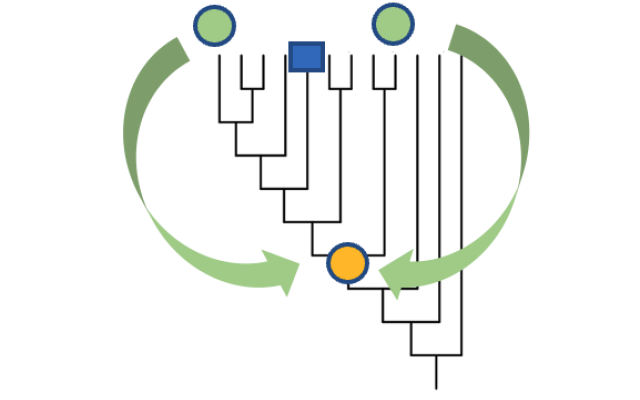
Zum ersten Mal wurden Biodaten von Menschen mit denen anderer Organismen integriert, um das bislang umfassendste Bild der menschlichen Genfunktion zu erstellen. Die neue Ressource „PAN-GO” nutzt ein von SIB-Wissenschaftlern mitentwickeltes Evolutionsmodell, um über 20.000 menschlichen Genen bekannte Funktionen zuzuordnen. An der Arbeit waren mehr als 150 Biologen beteiligt, die zum internationalen Gene Ontology Consortium beitragen. Die Ergebnisse werden heute in der Fachzeitschrift Nature veröffentlicht.
Innovative Bioinformatik schafft neue biomedizinische Ressource
Forscher, die biomedizinische Big-Data-Studien durchführen, können nun genauere und aufschlussreichere Einblicke in menschliche Krankheiten, Zellbiologie und vieles mehr gewinnen. Die PAN-GO-Funktionsdatenbank schließt Lücken in den experimentellen Daten zu menschlichen Genfunktionen, indem sie Daten aus verwandten Genen in Modellorganismen – darunter Mäuse, Zebrafische, Fruchtfliegen, Hefen und sogar Pflanzen – integriert. Die neue Datenbank ist für alle frei zugänglich und in einem maschinenlesbaren Format strukturiert, das künstliche Intelligenz und andere computergestützte Datenanalysen ermöglicht.
PAN-GO ist Teil der Gene Ontology (GO) Wissensdatenbank, die vom Gene Ontology Consortium entwickelt wurde, um umfassende und aktuelle Informationen über Genfunktionen im gesamten Stammbaum des Lebens bereitzustellen. GO wird vom NIH finanziert und aufgrund seiner Verwendung bei der Analyse und Interpretation von Biodaten jährlich in über 30 000 Publikationen zitiert. Wissenschaftler der Swiss-Prot-Gruppe des SIB sind Mitglieder des Konsortiums und tragen seit seiner Gründung vor 25 Jahren zu GO bei.
Die neue Ressource wurde von der Swiss-Prot-Gruppe der SIB, der Keck School of Medicine der University of Southern California (USC) und anderen Institutionen entwickelt und kombiniert Fachwissen zur Extraktion artspezifischer Informationen über Proteinfunktionen aus der wissenschaftlichen Literatur mit der Verallgemeinerung dieser Informationen über Arten hinweg durch modernste, groß angelegte Evolutionsmodellierung. Dank dieses innovativen Ansatzes können nun über 20.000 oder 82 % der menschlichen proteinkodierenden Gene bekannten Funktionen zugeordnet werden.
PAN-GO trägt zur Arbeit von Swiss-Prot bei, maschinenlesbares Wissen über Biologie zu generieren, und ergänzt UniProt, die führende Protein-Knowledgebase, die von der Gruppe mitentwickelt wurde. Seine Entwicklung steht im Einklang mit der Mission der SIB, die Grenzen der Datenwissenschaft zu erweitern, Innovationen in Medizin und Biodiversität zu beschleunigen und sicherzustellen, dass biologisches Wissen zum Nutzen von Wissenschaft und Gesellschaft allgemein zugänglich ist.
PAN-GO ist Teil der Gene Ontology (GO) Wissensdatenbank, die vom Gene Ontology Consortium entwickelt wurde, um umfassende und aktuelle Informationen über Genfunktionen im gesamten Stammbaum des Lebens bereitzustellen. GO wird vom NIH finanziert und aufgrund seiner Verwendung bei der Analyse und Interpretation von Biodaten jährlich in über 30 000 Publikationen zitiert. Wissenschaftler der Swiss-Prot-Gruppe des SIB sind Mitglieder des Konsortiums und tragen seit seiner Gründung vor 25 Jahren zu GO bei.
Eine Fülle neuer Informationen aus der Evolutionsmodellierung
Die Funktion von Genen wurde traditionell auf der Ebene eines bestimmten Gens in einer bestimmten Spezies und auf zwei Arten bestimmt: anhand experimenteller Daten und anhand computergestützter Vorhersagen. Die Evolutionsmodellierung von PAN-GO bietet eine leistungsstarke dritte Methode, mit der mehr als doppelt so viele funktionelle Merkmale für menschliche Gene identifiziert werden können als derzeit durch kuratierte experimentelle Daten zu menschlichen Genen verfügbar sind, und etwa dreimal so viele wie durch computergestützte Tools vorhergesagt werden.
Neue biologische Erkenntnisse und experimentelle Richtungen
Die Entwickler von PAN-GO haben gezeigt, dass die Ressource klarere und informativere Erkenntnisse liefert als bisher verfügbare computergestützte Genomanalysen – beispielsweise beim Vergleich von Genen, die in einem bestimmten Krebszelltyp exprimiert werden, mit den entsprechenden normalen Zelltypen.
Die Evolutionsmodelle selbst können verwendet werden, um zu untersuchen, wie und wann verschiedene Genfunktionen entstanden sind. Eine erste Analyse zeigt, dass die meisten menschlichen Gene seit Hunderten von Millionen Jahren oder länger dieselbe Funktion erfüllen, sogar schon bevor unsere Vorfahren Tiere waren.
PAN-GO wird auch als Leitfaden für die zukünftige Forschung zu den rund 3.600 menschlichen proteinkodierenden Genen dienen, deren biologische Funktion noch unbekannt ist, sowie zu den Tausenden weiteren Genen, deren Funktionen nur teilweise bekannt sind. Forscher können über die Website Vorschläge zur Aktualisierung der Ressource einreichen und so zu ihrer kontinuierlichen Verbesserung beitragen.
PAN-GO und UniProt: komplementär und von gegenseitigem Nutzen
Die zur Erstellung von PAN-GO verwendeten Evolutionsmodelle wurden anhand von Referenzproteinsätzen (Proteomen) in UniProt für verschiedene Spezies erstellt. Die Modelle nutzten auch funktionale Annotationen in UniProt und anderen Datenbanken, die von Mitgliedern des GO-Konsortiums erstellt wurden, d. h. experimentelle Nachweise zur Proteinfunktion, die in der wissenschaftlichen Literatur von erfahrenen Biokuratoren identifiziert und mit der entsprechenden Proteinsequenz in den Datenbanken versehen wurden.
Geneinträge in PAN-GO und der umfassenderen GO-Wissensdatenbank sind mit den entsprechenden Proteinträgen in UniProt verknüpft, und funktionale Annotationen in PAN-GO werden in UniProt importiert. Die neuen Annotationen ermöglichen es den Biokuratoren des SIB auch, in der Literatur nach experimentellen Daten zu suchen, um diese evolutionär abgeleiteten Funktionen zu bestätigen. Diese komplementäre Beziehung erhöht den Wert von UniProt als äußerst zuverlässige Quelle für die neuesten wissenschaftlichen Erkenntnisse über Proteine.
Reference(s)
Feuermann, M., Mi, H., Gaudet, P. et al. Ein Kompendium menschlicher Genfunktionen, abgeleitet aus evolutionären Modellen. Nature (2025).
Bild: Adaptiert aus Extended Data Figure 1 aus dem Artikel