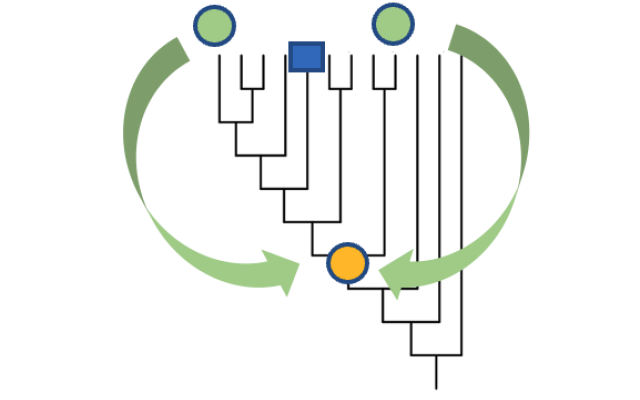
Per la prima volta, i dati biologici relativi agli esseri umani sono stati integrati con quelli di altri organismi per fornire il quadro più completo mai realizzato fino ad oggi delle funzioni dei geni umani. La nuova risorsa "PAN-GO" ha utilizzato modelli evolutivi sviluppati in collaborazione con gli scienziati del SIB per assegnare funzioni note a oltre 20.000 geni umani. Il lavoro ha coinvolto più di 150 biologi che hanno contribuito al Gene Ontology Consortium internazionale ed è stato pubblicato oggi sulla rivista Nature.
La bioinformatica innovativa crea una nuova risorsa biomedica
I ricercatori che conducono studi biomedici sui big data possono ora ottenere informazioni più accurate e approfondite sulle malattie umane, sulla biologia cellulare e altro ancora. La risorsa PAN-GO functionome colma le lacune nei dati sperimentali sulle funzioni dei geni umani, integrando i dati provenienti da geni correlati in organismi modello, tra cui topi, zebrafish, moscerini della frutta, lieviti e persino piante. La nuova risorsa è accessibile a tutti e strutturata in un formato leggibile da computer che consente l'intelligenza artificiale e altre analisi computazionali dei dati.
PAN-GO fa parte della banca dati Gene Ontology (GO) sviluppata dal Gene Ontology Consortium, che lavora per fornire informazioni complete e aggiornate sulla funzione dei geni nell'albero della vita. Finanziato dal NIH, GO è citato ogni anno in oltre 30.000 pubblicazioni per il suo utilizzo nell'analisi e nell'interpretazione dei dati biologici. Gli scienziati del gruppo Swiss-Prot di Il SIB sono membri del consorzio e contribuiscono a GO sin dalla sua fondazione, avvenuta 25 anni fa.
Sviluppata dal gruppo Swiss-Prot di Il SIB, dalla Keck School of Medicine dell'Università della California del Sud (USC) e da altre istituzioni, la nuova risorsa combina le competenze nell'estrazione di informazioni specifiche sulle funzioni delle proteine dalle pubblicazioni scientifiche e nella generalizzazione di tali informazioni tra le specie attraverso modelli evolutivi all'avanguardia e su larga scala. Grazie a questo approccio innovativo, le funzioni note sono ora assegnate a oltre 20.000, ovvero all'82%, dei geni umani codificanti proteine.
PAN-GO contribuisce al lavoro di Swiss-Prot volto a generare conoscenze di biologia leggibili da computer e integra UniProt, la principale banca dati sulle proteine sviluppata in collaborazione dal gruppo. Il suo sviluppo è in linea con la missione di Il SIB di ampliare i confini della scienza dei dati, accelerare l'innovazione in campo medico e della biodiversità e garantire che le conoscenze biologiche siano ampiamente accessibili a beneficio della scienza e della società.
PAN-GO fa parte della banca dati Gene Ontology (GO) sviluppata dal Gene Ontology Consortium, che lavora per fornire informazioni complete e aggiornate sulla funzione dei geni nell'albero della vita. Finanziato dal NIH, GO è citato ogni anno in oltre 30.000 pubblicazioni per il suo utilizzo nell'analisi e nell'interpretazione dei dati biologici. Gli scienziati del gruppo Swiss-Prot di Il SIB sono membri del consorzio e contribuiscono a GO sin dalla sua fondazione, avvenuta 25 anni fa.
Una ricchezza di nuove informazioni dalla modellizzazione evolutiva
La funzione dei geni è stata tradizionalmente determinata a livello di un gene specifico in una specie specifica, e in uno dei due modi seguenti: dati sperimentali e previsioni computazionali. La modellizzazione evolutiva di PAN-GO fornisce un potente terzo metodo, identificando più del doppio delle caratteristiche funzionali dei geni umani rispetto a quelle attualmente disponibili attraverso dati sperimentali curati sui geni umani, e circa il triplo rispetto a quelle previste dagli strumenti computazionali.
Nuove scoperte biologiche e direzioni sperimentali
Gli sviluppatori di PAN-GO hanno dimostrato che questa risorsa genera informazioni più chiare e dettagliate rispetto a quelle precedentemente disponibili dalle analisi genomiche computazionali, ad esempio quando si confrontano i geni espressi in un tipo specifico di cellula cancerosa con il corrispondente tipo di cellula normale.
I modelli evolutivi stessi possono essere utilizzati per esaminare come e quando sono sorte le diverse funzioni geniche. Da una prima analisi emerge che la maggior parte dei geni umani ha svolto la stessa funzione per centinaia di milioni di anni o più, anche prima che i nostri antenati fossero animali.
PAN-GO contribuirà anche a guidare la ricerca futura sui circa 3.600 geni umani che codificano le proteine, la cui funzione biologica rimane sconosciuta, così come sulle migliaia di altri geni le cui funzioni sono solo parzialmente note. I ricercatori possono inviare suggerimenti per l'aggiornamento della risorsa attraverso il suo sito web, contribuendo a garantire continui miglioramenti nel tempo.
PAN-GO e UniProt: complementari e reciprocamente vantaggiosi
I modelli evolutivi utilizzati per creare PAN-GO sono stati costruiti utilizzando set di proteine di riferimento (proteomi) presenti in UniProt per diverse specie. I modelli hanno inoltre sfruttato le annotazioni funzionali presenti in UniProt e in altri database creati dai membri del GO Consortium, ovvero prove sperimentali sulla funzione delle proteine identificate nella letteratura scientifica da biocuratori esperti e fornite con la corrispondente sequenza proteica nei database.
Le voci relative ai geni presenti su PAN-GO e nella più ampia banca dati GO sono collegate alle voci relative alle proteine corrispondenti presenti su UniProt, mentre le annotazioni funzionali presenti su PAN-GO vengono importate su UniProt. Le nuove annotazioni consentono inoltre ai biocuratori al SIB di cercare nella letteratura dati sperimentali che confermino queste funzioni dedotte dal punto di vista evolutivo. Questa relazione complementare aumenta il valore di UniProt come fonte altamente affidabile delle più recenti conoscenze scientifiche sulle proteine.
Reference(s)
Feuermann, M., Mi, H., Gaudet, P. et al. Una raccolta delle funzioni dei geni umani derivata dalla modellizzazione evolutiva. Nature (2025).
Immagine: Adattato dalla Figura 1 dei dati estesi dell'articolo