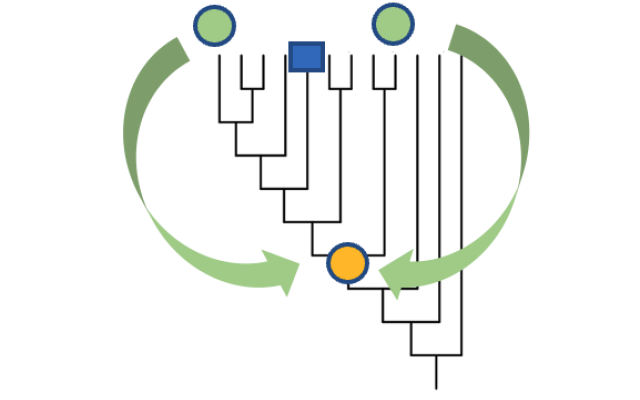
Pour la première fois, des données biologiques humaines ont été intégrées à celles d'autres organismes afin de fournir l'image la plus complète à ce jour de la fonction des gènes humains. La nouvelle ressource « PAN-GO » a utilisé une modélisation évolutive développée conjointement par des scientifiques du SIB pour attribuer des fonctions connues à plus de 20 000 gènes humains. Ce travail, auquel ont contribué plus de 150 biologistes du Gene Ontology Consortium international, est publié aujourd'hui dans la revue Nature.
La bioinformatique innovante crée une nouvelle ressource biomédicale
Les chercheurs qui mènent des études biomédicales sur le big data peuvent désormais obtenir des informations plus précises et plus instructives sur les maladies humaines, la biologie cellulaire et bien d'autres domaines. La ressource fonctionnelle PAN-GO comble les lacunes des données expérimentales sur les fonctions des gènes humains en intégrant des données provenant de gènes apparentés chez des organismes modèles, notamment des souris, des poissons zèbres, des mouches à fruits, des levures et même des plantes. Cette nouvelle ressource est accessible à tous et structurée dans un format lisible par machine qui permet l'intelligence artificielle et d'autres analyses de données computationnelles.
PAN-GO fait partie de la base de connaissances Gene Ontology (GO) développée par le Gene Ontology Consortium, qui s'efforce de fournir des informations complètes et actualisées sur la fonction des gènes dans l'arbre de la vie. Financé par le NIH, GO est cité chaque année dans plus de 30 000 publications pour son utilisation dans l'analyse et l'interprétation des données biologiques. Les scientifiques du groupe Swiss-Prot du SIB sont membres du consortium et contribuent à GO depuis sa création il y a 25 ans.
Développée par le groupe Swiss-Prot du SIB, la Keck School of Medicine de l'Université de Californie du Sud (USC) et d'autres institutions, cette nouvelle ressource combine l'expertise en matière d'extraction d'informations spécifiques à une espèce sur la fonction des protéines à partir de la littérature scientifique et la généralisation de ces informations à toutes les espèces grâce à une modélisation évolutive de pointe à grande échelle. Grâce à cette approche innovante, des fonctions connues sont désormais attribuées à plus de 20 000 gènes codant pour des protéines humaines, soit 82 % d'entre eux.
PAN-GO contribue au travail de Swiss-Prot visant à générer des connaissances biologiques lisibles par machine et complète UniProt, la principale base de connaissances sur les protéines co-développée par le groupe. Son développement s'inscrit dans la mission du SIB qui consiste à repousser les limites de la science des données, à accélérer l'innovation dans les domaines de la médecine et de la biodiversité, et à garantir que les connaissances biologiques soient largement accessibles au profit de la science et de la société.
PAN-GO fait partie de la base de connaissances Gene Ontology (GO) développée par le Gene Ontology Consortium, qui s'efforce de fournir des informations complètes et actualisées sur la fonction des gènes dans l'arbre de la vie. Financé par le NIH, GO est cité chaque année dans plus de 30 000 publications pour son utilisation dans l'analyse et l'interprétation des données biologiques. Les scientifiques du groupe Swiss-Prot du SIB sont membres du consortium et contribuent à GO depuis sa création il y a 25 ans.
Une mine d'informations nouvelles issues de la modélisation évolutive
La fonction des gènes a traditionnellement été déterminée au niveau d'un gène spécifique chez une espèce spécifique, et ce de deux manières : à partir de données expérimentales ou de prédictions informatiques. La modélisation évolutive de PAN-GO offre une troisième méthode puissante, qui permet d'identifier plus de deux fois plus de caractéristiques fonctionnelles des gènes humains que les données expérimentales actuellement disponibles sur les gènes humains, et environ trois fois plus que les prédictions des outils informatiques.
Nouvelles perspectives biologiques et orientations expérimentales
Les développeurs de PAN-GO ont démontré que cette ressource génère des informations plus claires et plus pertinentes que celles obtenues précédemment à partir d'analyses génomiques computationnelles, par exemple lors de la comparaison de gènes exprimés dans un type spécifique de cellule cancéreuse avec le type de cellule normale correspondant.
Les modèles évolutifs eux-mêmes peuvent être utilisés pour examiner comment et quand différentes fonctions génétiques sont apparues. Une première analyse montre que la plupart des gènes humains ont rempli la même fonction pendant des centaines de millions d'années, voire avant que nos ancêtres ne soient encore des animaux.
PAN-GO contribuera également à orienter les recherches futures sur les quelque 3 600 gènes humains codant pour des protéines dont la fonction biologique reste inconnue, ainsi que sur les milliers d'autres dont les fonctions ne sont que partiellement connues. Les chercheurs peuvent soumettre des suggestions pour mettre à jour cette ressource via son site web, contribuant ainsi à son amélioration continue au fil du temps.
PAN-GO et UniProt : complémentaires et mutuellement bénéfiques
Les modèles évolutifs utilisés pour créer PAN-GO ont été construits à partir d'ensembles de protéines de référence (protéomes) dans UniProt pour différentes espèces. Les modèles ont également exploité les annotations fonctionnelles dans UniProt et d'autres bases de données créées par les membres du consortium GO, c'est-à-dire des preuves expérimentales sur la fonction des protéines identifiées dans la littérature scientifique par des biocurateurs experts et fournies avec la séquence protéique correspondante dans les bases de données.
Les entrées de gènes dans PAN-GO et la base de connaissances GO plus large renvoient aux entrées de protéines correspondantes dans UniProt, et les annotations fonctionnelles dans PAN-GO sont importées dans UniProt. Les nouvelles annotations permettent également aux biocurateurs du SIB de rechercher dans la littérature des données expérimentales pour confirmer ces fonctions déduites de l'évolution. Cette relation complémentaire renforce la valeur d'UniProt en tant que source hautement fiable des dernières connaissances scientifiques sur les protéines.
Reference(s)
Feuermann, M., Mi, H., Gaudet, P. et al. A compendium of human gene functions derived from evolutionary modelling. Nature (2025).
Image : Adapté de la figure 1 des données supplémentaires de l'article