Jüngste Fortschritte im Kampf gegen Typ-2-Diabetes (T2D) sind das Ergebnis eines europaweiten Kooperationsprojekts namens IMIDIA. SIB war sowohl für die Koordinierung der umfangreichen Patientendaten (als Data Coordination Centre, DCC) als auch für die Analyse der Daten zur Ermittlung von Biomarkern für die Früherkennung der Krankheit verantwortlich. Zu den wichtigsten Ergebnissen der Forschung gehört die Entdeckung von Molekülen, die als Biomarker dienen könnten, um die Krankheit bis zu neun Jahre vor ihrer Diagnose zu erkennen. Diese Ergebnisse wurden in zwei Publikationen zusammengefasst, die den erfolgreichen Abschluss des IMIDIA-Projekts markieren – einer öffentlich-privaten Partnerschaft der Innovative Medicines Initiative (IMI) unter der Leitung von Bernard Thorens von der Universität Lausanne (UNIL). Die SIB wurde über ihre Gruppen Vital-IT (Computational Biology) und Swiss-Prot (Expert Biocuration) inzwischen als DCC für zwei weitere IMI-Projekte zur Bekämpfung von Diabetes ausgewählt und entwickelt sich zu einem Referenzpartner für Bioinformatik in europäischen Gesundheitsprogrammen.
Von Mäusen und Menschen – und Diabetes: Zwei Studien zeigen neue Marker für T2D
Es gibt zwar therapeutische Möglichkeiten zur Behandlung von Diabetes, aber keine zur Vorbeugung oder Heilung der Krankheit. Dies veranlasste Forscher dazu, 1) die molekularen Funktionsstörungen, die der Krankheit zugrunde liegen, zu verstehen und 2) zirkulierende Biomarker zu identifizieren, die die Anfälligkeit einer Person für T2D vorhersagen könnten.
In einer ersten Studie, die von Mark Ibberson, Seniorautor der Vital-IT-Gruppe der SIB, koordiniert und in Molecular Metabolism veröffentlicht wurde, identifizierte das Team ein Schlüsselgen, das mit T2D in Verbindung steht. Mithilfe einer Netzwerkanalyse, die transkriptomische und phänotypische Daten integrierte, schien das Gen Elovl2 mit der Insulinsekretion bei Mäusen in Zusammenhang zu stehen. Der Befund wurde in menschlichen Betazelllinien der Bauchspeicheldrüse bestätigt, d. h. in Zellen, die denen ähneln, die bei T2D betroffen sind.
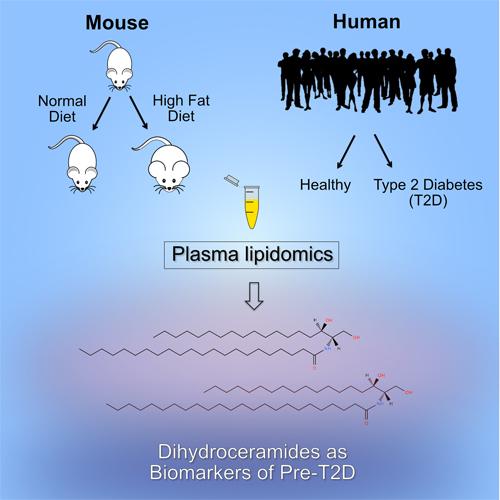
In einer parallelen Studie unter der Leitung von Bernard Thorens (UNIL) mit Leonore Wigger von der Vital-IT-Gruppe der SIB als Hauptautorin, die in Cell Reports veröffentlicht wurde, identifizierten die Forscher mehrere Lipide als potenzielle frühe Biomarker für T2D. Auf eine vorläufige Lipidomik-Studie an Mäusen, die viele Lipidklassen umfasste, folgte eine gezielte Analyse an zwei menschlichen Kohorten aus der Schweiz (CoLAUS) und Frankreich (DESIR). Eine bestimmte Lipidklasse, die Dihydroceramide, war in den Gruppen der Studienteilnehmer, die sich auf Diabetes zubewegten, bis zu neun Jahre vor der Diagnose durchweg erhöht.
An diesen Studien waren akademische Teams, Pharmaunternehmen und ein kleines bis mittleres Unternehmen (KMU) beteiligt. Darüber hinaus wurden die Ergebnisse aus groß angelegten Experimenten an Mäusen anhand von Kohorten menschlicher Patienten, die im Rahmen des Projekts Innovative Medicines Initiative for Diabetes (IMIDIA) zur Verfügung gestellt wurden, kreuzvalidiert. Die Ergebnisse unterstreichen daher die wichtige Rolle öffentlich-privater Partnerschaften wie der IMI – Europas größter öffentlich-privater Initiative – für solche Fortschritte und die Verbesserung der öffentlichen Gesundheit.
SIB, ein Schweizer Kompetenzzentrum für Bioinformatik und Gesundheitsdaten
Über diese wissenschaftlichen Erkenntnisse und ihre potenziellen klinischen Anwendungen hinaus zeigt die Rolle von SIB im Rahmen von IMIDIA den einzigartigen Wert und die Fähigkeiten des Instituts als Kompetenzzentrum im Kontext groß angelegter Gesundheitsdatenprojekte.
Tatsächlich stehen solche Projekte vor großen Herausforderungen, wie z. B.: die Interoperabilität der Patientendaten unter Wahrung ihrer Sicherheit zu gewährleisten; innovative Modellierungsansätze zur Analyse der Daten zu entwickeln; diese Analysen mit entsprechender Rechenleistung zu unterstützen; die Ergebnisse mit kuratierten Datenbanken zu verknüpfen.
Die SIB vereint unter einem Dach Know-how in den Bereichen Bioinformatik, Hochleistungsrechnen (z. B. die Vital-IT-Gruppe) sowie führende Expertise in der Biokuration (z. B. die Swiss-Prot-Gruppe, die die weltweit meistgenutzte Proteininformationsquelle unterhält). Das IMIDIA-Projekt stützte sich insbesondere auf die SwissLipids-Wissensdatenbank, die von Swiss-Prot in Zusammenarbeit mit der Schweizer Initiative für Systembiologie (SystemsX.ch) entwickelt wurde. Diese umfassende Bibliothek mit über 300'000 bekannten und theoretisch möglichen Lipidstrukturen wird durch von Experten kuratierte Informationen zum Lipidstoffwechsel, zu Proteininteraktionen und zum Vorkommen in Organellen, Zellen, Geweben und Organen ergänzt.
Neben ihrer Datenwissenschaftskompetenz kann die SIB dank ihrer einzigartigen Position an der Schnittstelle zwischen öffentlichem und privatem Sektor nahtlos mit Partnern aus Wissenschaft und Industrie zusammenarbeiten, um die Forschung zu beschleunigen und in Vorteile für die menschliche Gesundheit umzusetzen.
Seit ihrer Beteiligung am IMIDIA-Projekt wurde die SIB aufgrund ihrer Rolle als Wegbereiterin im Bereich der Lebenswissenschaften, sowohl in der Datenkuratierung als auch in der Bioinformatik, als DCC für Folgeprojekte ausgewählt: RHAPSODY und BEAt-DKD. Während IMIDIA und RHAPSODY, beide unter der Leitung von Bernard Thorens an der Universität Lausanne, sich auf die Identifizierung neuer Biomarker für T2D konzentrieren, konzentriert sich BEAt-DKD auf die Suche nach Biomarkern für die diabetische Nierenerkrankung (DKD), die häufigste Ursache für Nierenversagen, von der bis zu 1 von 4 Menschen mit Diabetes betroffen sind. SIB ist der einzige Schweizer Vertreter in diesem Projekt.
Vollständige Medienmitteilung (Englisch – Französisch – Deutsch)
Reference(s)
Cruciani-Guglielmacci C et al. Die molekulare Phänotypisierung mehrerer Mausstämme unter metabolischer Belastung deckt eine Rolle von Elovl2 bei der glukoseinduzierten Insulinsekretion auf. Molecular Metabolism 2017
Wigger L et al. Plasma-Dihydroceramide sind Kandidaten für Biomarker für die Anfälligkeit für Diabetes bei Mäusen und Menschen. Cell Reports 2017;18: 2269 – 2279