Eine internationale Studie zeigt die beispiellose Fähigkeit des Pneumocystis-Pilzes, das menschliche Immunsystem zu überlisten. Philippe Hauser vom Universitätsspital Lausanne (CHUV) leitete die Arbeiten, und die Einzigartigkeit der molekularen Mechanismen wurde anhand von Bioinformatik-Analysen unter der Leitung von Marco Pagni von der SIB aufgedeckt. Die Ergebnisse sind in Nature Communications veröffentlicht.
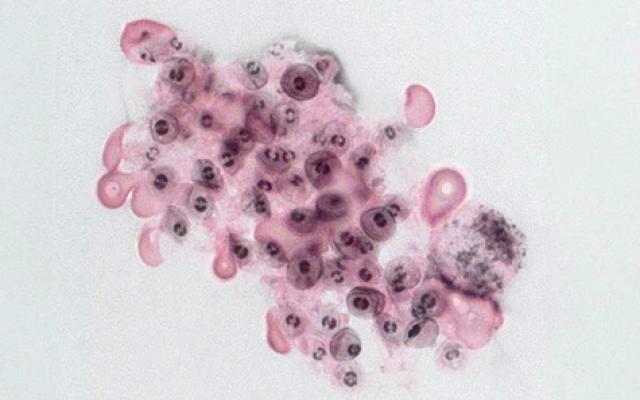
Mikrobielle Parasiten, die beim Menschen schwere Krankheiten wie Malaria oder Schlafkrankheit verursachen, müssen das Immunsystem umgehen, um sich im menschlichen Körper vermehren zu können. Das Labor von Philippe Hauser (CHUV) hat in Zusammenarbeit mit Marco Pagni von der SIB und anderen wichtigen Partnern die Mechanismen untersucht, mit denen der Pneumocystis-Pilz dies erreicht, der beispielsweise bei immungeschwächten Patienten, Organtransplantierten oder Blutkrebspatienten tödliche Lungenentzündungen verursacht.
Ein neuer molekularer Mechanismus der Antigenvariation entdeckt
Die in der Fachzeitschrift Nature Communications veröffentlichte Studie zeigt, dass dieser Erreger durch zufällige Neukombination Tausender seiner Gene, die in seinen weltweiten Populationen vorkommen, in jedem Stamm einen neuen Satz von 80 Genen bildet. Innerhalb dieses Satzes wird dann pro einzelne Zelle des Pilzes ein einziges Gen exprimiert, um seine Oberfläche mit einer „Hülle” zu überziehen, die das Immunsystem nicht erkennen und bekämpfen kann, da es ihr in der Regel noch nie begegnet ist. Dieser molekulare Mechanismus der Antigenvariation auf Zellebene war bisher nicht bekannt und ergänzt bereits bekannte Mechanismen, beispielsweise beim Trypanosom, das die Schlafkrankheit verursacht. Die einzelnen Zellen des Pilzes wechseln dann häufig ihre „Hülle”, um der Erkennung durch das Immunsystem zu entgehen, das sich während der Infektion entwickelt. Das Verständnis dieser einzigartigen Tarnmechanismen könnte zu einer neuen therapeutischen Strategie zur Bekämpfung der Krankheit führen.
Aufbauend auf 15 Jahren Forschung
Diese Entdeckung ist das Ergebnis von fünfzehn Jahren Forschung an diesem Erreger, bei der das Team von Philippe Hauser eng mit Marco Pagni, Teamleiter Computational Biology bei der SIB-Gruppe Vital-IT, für den Teil Bioinformatik zusammengearbeitet hat. Für die aktuelle Studie entwickelte das SIB ein wichtiges Computerverfahren, mit dem die Gene, aus denen sich jeder Stamm des Pilzes zusammensetzt, unterschieden werden können.
„Pneumocystis ist ein sehr schwieriger Erreger. Da er nicht in vitro kultiviert werden kann, müssen wir mit weniger mehr sagen können, und hier kommt die Bioinformatik ins Spiel. Während unserer langjährigen Forschungszusammenarbeit mit dem Labor von Philippe Hauser haben wir zunächst das Genom sequenziert und uns dann auf bestimmte Regionen konzentriert, die sehr kompliziert zu assemblieren sind – und das alles aus wenigen wertvollen klinischen Proben”, sagt Marco Pagni. „Es ist daher fantastisch, dass wir heute einen neuen Mechanismus dokumentieren können, der erklärt, wie der Erreger unser Immunsystem umgeht.”
Die Studie ist das Ergebnis einer internationalen Zusammenarbeit von Forschern der Universität Lissabon, des CHU in Brest, der Universität Cincinnati und des Instituts für Biomedizin in Sevilla sowie des Instituts für Infektionskrankheiten der Universität Bern in der Schweiz.
Reference(s)
Meier C S et al. Fungal antigenic variation using mosaicism and reassortment of subtelomeric genes’ repertoires, Nature Communications 2023.
Bannerbild: Pneumocystis. Bildnachweis: Philippe Hauser, CHUV