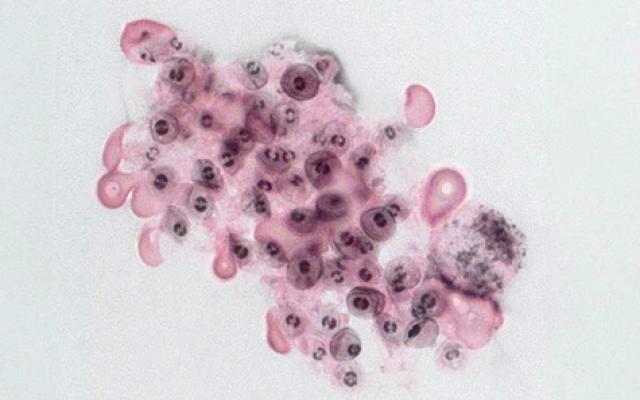
Une étude internationale montre la capacité sans précédent du champignon Pneumocystis à déjouer le système immunitaire humain. Philippe Hauser, de l'Hôpital universitaire de Lausanne (CHUV), a dirigé ces travaux, et le caractère unique des mécanismes moléculaires impliqués a été révélé grâce à des analyses bioinformatiques menées par Marco Pagni au SIB. Les résultats sont publiés dans Nature Communications.
Les parasites microbiens responsables de maladies graves chez l'homme, telles que le paludisme ou la maladie du sommeil, doivent échapper au système immunitaire pour se multiplier dans l'organisme humain. Le laboratoire de Philippe Hauser (CHUV) collabore avec Marco Pagni du SIB et d'autres partenaires clés pour étudier les mécanismes utilisés à cette fin par le champignon Pneumocystis, qui provoque une pneumonie mortelle chez les patients immunodéprimés, ceux qui ont subi une transplantation d'organe ou ceux qui souffrent d'un cancer du sang, par exemple.
Un nouveau mécanisme moléculaire de variation antigénique révélé
L'étude, publiée dans la revue Nature Communications, révèle que cet agent pathogène utilise un brassage aléatoire de milliers de ses gènes présents dans ses populations mondiales pour créer un nouvel ensemble de 80 gènes dans chaque souche. Au sein de cet ensemble, un seul gène est ensuite exprimé par cellule individuelle du champignon, afin de recouvrir sa surface d'un « manteau » que le système immunitaire ne peut pas reconnaître et combattre, car il ne l'a généralement jamais rencontré auparavant. Ce mécanisme moléculaire de variation antigénique au niveau cellulaire était jusqu'à présent inconnu et s'ajoute à ceux déjà connus, par exemple chez le trypanosome responsable de la maladie du sommeil. Les cellules individuelles du champignon changent ensuite fréquemment de « manteau » pour éviter d'être reconnues par le système immunitaire qui se développe au cours de l'infection. La compréhension de ces mécanismes de camouflage uniques pourrait conduire à une nouvelle stratégie thérapeutique pour lutter contre la maladie.
S'appuyant sur 15 ans de recherche
Cette découverte est l'aboutissement de quinze années de recherche sur ce pathogène, durant lesquelles l'équipe de Philippe Hauser a travaillé en étroite collaboration avec Marco Pagni, Team Lead Computational Biology au sein du groupe Vital-IT du SIB, pour la partie bioinformatique. Pour la présente étude, le SIB a développé une procédure informatique essentielle permettant de différencier les gènes constituant l'ensemble présent dans chaque souche du champignon.
“Pneumocystis est un agent pathogène très difficile à étudier. Comme il ne peut pas être cultivé in vitro, nous devons être capables d'en dire plus avec moins, et c'est là que la bioinformatique entre en jeu. Au cours de notre longue collaboration avec le laboratoire de Philippe Hauser, nous avons commencé par séquencer son génome, puis nous nous sommes concentrés sur des régions spécifiques très complexes à assembler, le tout à partir de quelques précieux échantillons cliniques », explique Marco Pagni. « C'est donc fantastique de pouvoir aujourd'hui documenter un nouveau mécanisme expliquant sa capacité à échapper à notre système immunitaire. »
Cette étude est le fruit d'une collaboration internationale entre des chercheurs de l'université de Lisbonne, du CHU de Brest, de l'université de Cincinnati et de l'Institut de biomédecine de Séville, ainsi que de l'Institut des maladies infectieuses de l'université de Berne en Suisse.
Reference(s)
Meier C S et al. Variation antigénique fongique utilisant le mosaïcisme et le réassortiment des répertoires de gènes subtélomériques, Nature Communications 2023.
Image de bannière : Pneumocystis. Crédit : Philippe Hauser, CHUV