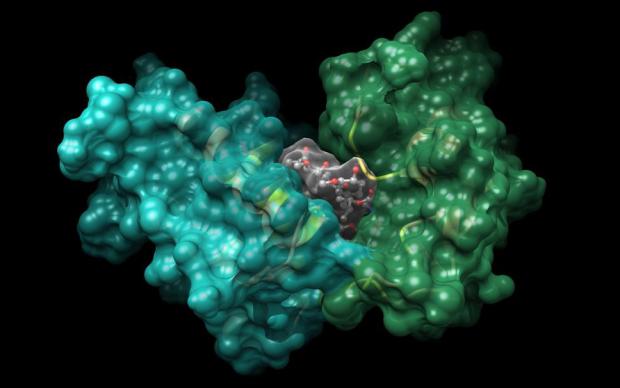
Die Molekulare Modellierungsgruppe untersucht molekulare Wechselwirkungen mithilfe von Modellierungstechniken wie Homologiemodellierung, Molekulardynamik, Docking und Freie-Energie-Simulationen. Unsere Haupttätigkeit besteht in der Entwicklung und Anwendung von Methoden für das computergestützte Protein-Engineering und Wirkstoffdesign. Insbesondere entwickeln wir neuartige Inhibitoren für wichtige Ziele in der Krebstherapie und entwerfen optimierte Proteine, wie z. B. T-Zell-Rezeptoren, für die Krebsimmuntherapie. Wir entwickeln und pflegen Web-Tools für das Wirkstoffdesign, wie SwissDock, SwissTargetPrediction und SwissADME. Außerdem betreiben wir die Proteinmodellierungsanlage der Universität Lausanne.
Die Gruppe entwickelt die SIB-Ressource SwissDrugDesign, eine Suite webbasierter Software-Tools für das computergestützte Wirkstoffdesign