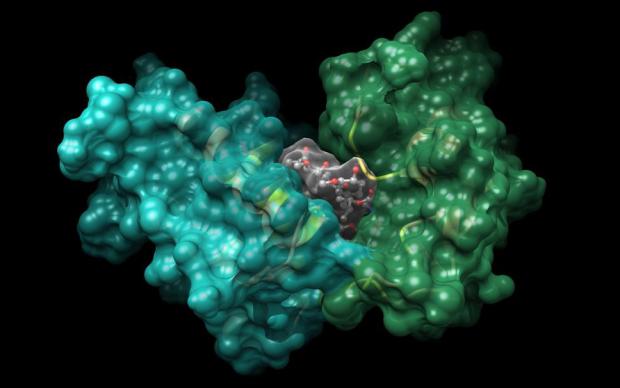
Le groupe de modélisation moléculaire étudie les interactions moléculaires à l'aide de techniques de modélisation telles que la modélisation par homologie, la dynamique moléculaire, l'ancrage et les simulations d'énergie libre. Notre activité principale consiste à développer et à appliquer des méthodes d'ingénierie protéique et de conception de médicaments assistées par ordinateur. Nous développons notamment de nouveaux inhibiteurs de cibles importantes pour le traitement du cancer et concevons des protéines optimisées, telles que des récepteurs de cellules T, pour l'immunothérapie du cancer. Nous développons et maintenons des outils web pour la conception de médicaments, tels que SwissDock, SwissTargetPrediction et SwissADME. Nous gérons également l'installation de modélisation des protéines de l'Université de Lausanne.
Le groupe développe la ressource SIB SwissDrugDesign, une suite d'outils logiciels web d'aide à la conception de médicaments