Des scientifiques du SIB et de l'Université de Bâle, ainsi que de Chelonia Applied Sciences (Bâle), participent à un effort européen visant à trouver des molécules actives contre le COVID-19 et à accélérer ce processus à l'avenir. Ils font partie d'Exscalate4CoV (E4C), un consortium public-privé regroupant 18 organisations de premier plan de l'UE, dont la plateforme européenne de supercalcul EXSCALATE, issues de 7 pays, qui a reçu 3 millions d'euros du programme Horizon 2020 de la Commission européenne. Les scientifiques utiliseront la ressource SWISS-MODEL du SIB pour générer des modèles 3D de protéines qui n'ont pas encore été élucidées expérimentalement, accélérant ainsi le criblage virtuel de médicaments potentiels.
Accélérer la recherche de médicaments contre la COVID-19
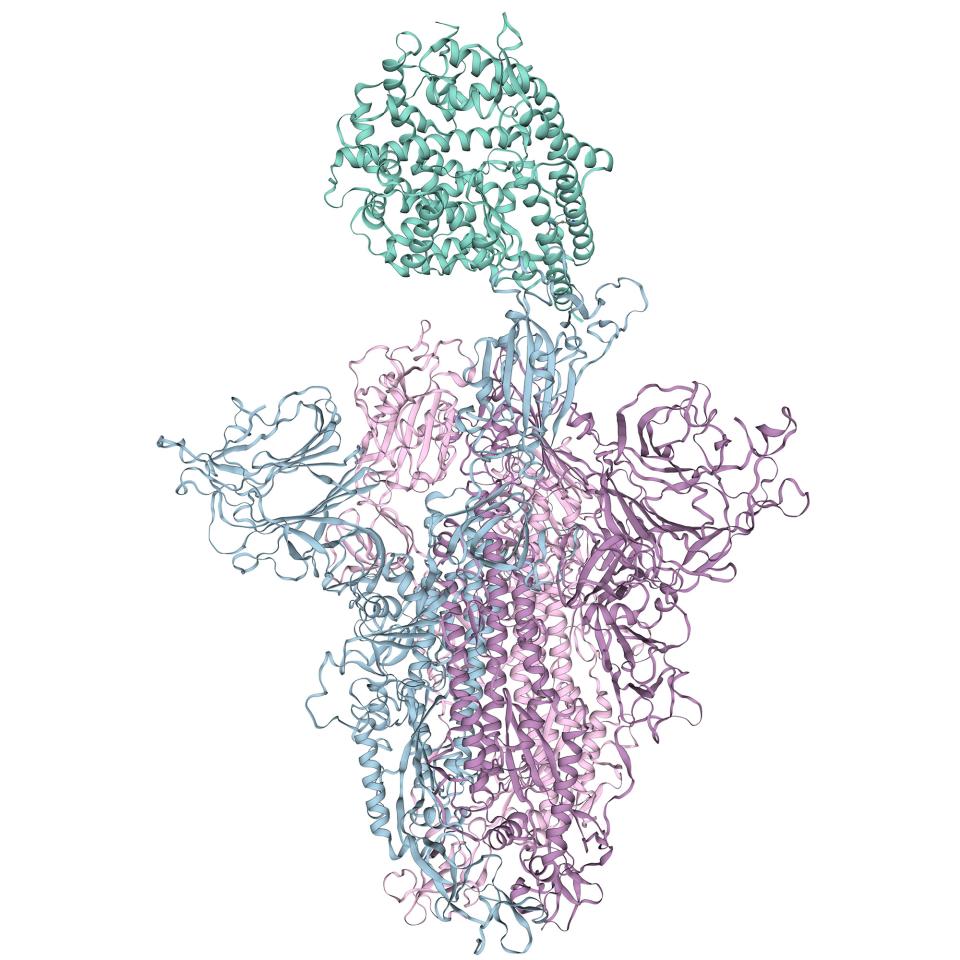
Dans le cadre du consortium E4C, le groupe SIB Computational Structural Biology (CSB) du Biozentrum (Université de Bâle) est chargé de générer des modèles tridimensionnels prédits des protéines COVID-19 in silico. « Comme il s'agit d'un virus relativement nouveau, les scientifiques n'ont commencé que récemment à caractériser expérimentalement les structures 3D des protéines virales. Les méthodes informatiques permettent de modéliser des structures à partir d'informations provenant de protéines apparentées, telles que celles du SARS-CoV, accélérant ainsi le processus de découverte de médicaments », explique Gerardo Tauriello, chef de l'équipe de développement au CSB. Cette approche est appelée « modélisation par homologie » et l'un des principaux outils pour la mettre en œuvre a été développé par l'équipe : le serveur SWISS-MODEL, une ressource libre d'accès du SIB. Les structures expérimentales et les modèles d'homologie seront utilisés par les autres participants à E4C pour identifier de petites molécules potentiellement capables de cibler le coronavirus. Ces molécules feront ensuite l'objet de tests supplémentaires dans le cadre d'expériencesin vitro, de modèles animaux et, en cas de succès, d'essais cliniques.
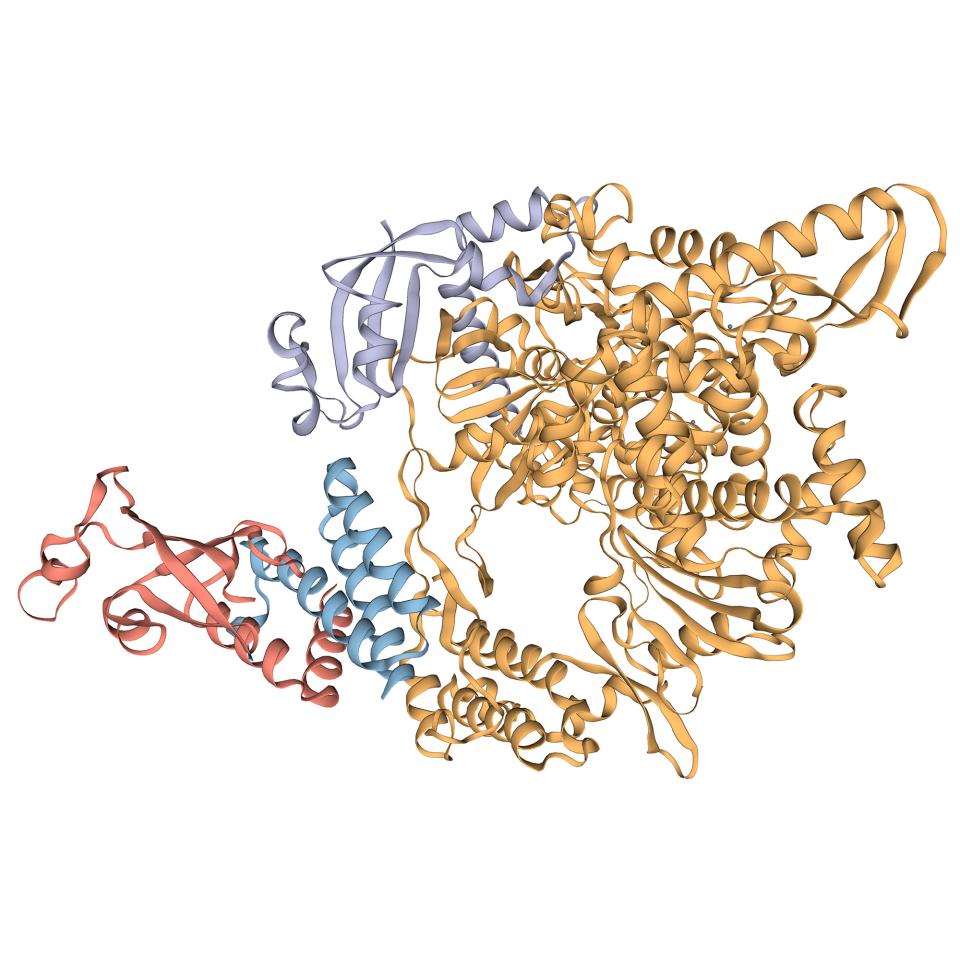
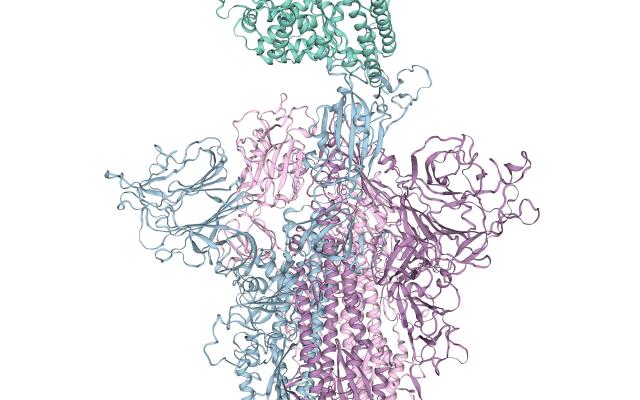
Examen minutieux des protéines en 3D pour faciliter la découverte de médicaments
Les virus, même s'ils ont besoin du mécanisme des cellules hôtes pour exprimer leur génome, dépendent des protéines codées dans leur génome, tout comme les organismes vivants. Chaque protéine possède une structure tridimensionnelle particulière, étroitement liée à sa fonction. Par exemple, une série de protéines transmembranaires sont utilisées par le virus pour se lier aux cellules hôtes. Les molécules médicamenteuses sont conçues pour se lier à des parties spécifiques d'une protéine afin de moduler sa fonction. Une représentation tridimensionnelle de la structure d'une protéine au niveau atomique est donc un aspect essentiel de la découverte rationnelle de médicaments.
Au-delà de la COVID-19 : accélérer la découverte de médicaments pour lutter contre les pandémies futures
E4C vise à tirer parti des enseignements et des avancées réalisés pendant la pandémie actuelle afin d'accélérer la découverte et l'identification de médicaments en cas de pandémies futures, en particulier lorsque l'agent pathogène est inconnu de la science et qu'il n'existe aucun traitement. « Aujourd'hui, le traitement d'un génome viral inconnu nécessite un travail manuel fastidieux de la part d'experts en modélisation, ce qui limite le temps de réaction. À l'avenir, dès qu'un nouveau virus sera séquencé, n'importe quel chercheur pourra utiliser SWISS-MODEL pour générer des modèles en quelques minutes et les rendre prêts à être utilisés et interprétés dans la plateforme EXSCALATE », explique Tauriello. Dans le cadre de l'E4C, le groupe CSB du SIB développera et évaluera un flux de travail automatisé et évolutif permettant de générer des modèles de protéines par homologie, directement à partir du génome de n'importe quel virus.
Coupler les meilleurs laboratoires de recherche en sciences de la vie avec une superinformatique puissante
E4C associe la plateforme européenne de supercalcul EXSCALATE (EXaSCale smArt pLatform Against paThogEns), capable d'exploiter une bibliothèque de 500 milliards de candidats médicaments grâce à une capacité de traitement de plus de 3 millions de molécules par seconde, à des laboratoires de recherche de pointe dans le domaine des sciences de la vie. Chelonia Applied Science, basée à Bâle, a conçu le consortium E4C et est chargée de ses activités de diffusion et d'exploitation. Silvano Coletti, directeur général de Chelonia, déclare : « La situation à laquelle le monde est confronté avec la pandémie de COVID-19 est sans précédent dans notre vie et nous touche tous, tant sur le plan professionnel que personnel. Exscalate4CoV est un effort informatique tout aussi sans précédent, qui vise à accélérer l'identification des médicaments contre la COVID-19 aujourd'hui et à devenir la plateforme de référence pour les pandémies de demain, y compris pour le processus de découverte et de développement dans la recherche pharmaceutique. »
Outre les membres suisses, le consortium E4C, coordonné par l'entreprise pharmaceutique italienne Dompé, comprend 16 institutions* issues de six pays. Pour en savoir plus, rendez-vous sur le site web EXSCALATE4CoV.
*Dompé Farmaceutici SPA, Politecnico di Milano (Département d'Électronique, d'Informatique et de Bioingénierie), Consorzio Interuniversitario CINECA (Innovation et applications en supercalcul), Université de Milan (Département des sciences pharmaceutiques), Université catholique de Louvain, Institut international de biologie moléculaire et cellulaire de Varsovie (LIMCB), Elettra Sincrotrone Trieste, Institut Fraunhofer pour la biologie moléculaire et l'écologie appliquée, Bsc Supercomputing Centre, Forschungszentrum Jülich, Université Federico II de Naples, Université de Cagliari, KTH Royal Institute of Technology (Département de physique appliquée), Associazione BigData, Istituto Nazionale di Fisica Nucleare (INFN) et Istituto nazionale per le malattie infettive Lazzaro Spallanzani.