Ein wissenschaftliches Kollektiv unter der Leitung des Earth BioGenome Project hat sich zum Ziel gesetzt, bis 2035 die Genome von 1,8 Millionen Tier-, Pflanzen- und anderen eukaryotischen Arten zu sequenzieren und zu assemblieren. Die Initiative wird Daten produzieren, die frei zugänglich sind, von öffentlichen Cloud-Infrastrukturen unterstützt werden und Forschern weltweit zur Verfügung stehen. Diese Referenzgenome sind entscheidend für die Entwicklung wirksamer Schutzstrategien und die Bekämpfung des Biodiversitätsverlusts. Das SIB bringt seine umfangreiche Erfahrung in der Datenwissenschaft ein, um die Entwicklung von Best Practices, die Bereitstellung der Recheninfrastruktur, die Verbreitung der Ergebnisse und das Training sicherzustellen.
Sequenzierung zur besseren Erhaltung der biologischen Vielfalt
Die biologische Vielfalt nimmt in den letzten Jahren vor allem aufgrund menschlicher Aktivitäten in alarmierendem Tempo ab. Die Erstellung des Referenzgenoms einer Spezies bildet die Grundlage für wertvolle Erkenntnisse über ihre Evolutionsgeschichte, genetische Vielfalt und einzigartigen Anpassungen. Diese Informationen sind entscheidend für die Entwicklung wirksamerer Schutzstrategien, beispielsweise für die Identifizierung genetisch relevanter Individuen für Zuchtprogramme, die Wiederherstellung der genetischen Vielfalt und das Verständnis der Anfälligkeit gegenüber Krankheiten und Umweltveränderungen.
Das Earth BioGenome Project (EBP) hat sich zum Ziel gesetzt, die Genome aller eukaryotischen Arten der Erde zu sequenzieren, zu katalogisieren und zu charakterisieren. Um die Genom-Assemblierungsprozesse zu optimieren und Best Practices zu entwickeln, haben sich zwei Genomprojekte – das Vertebrate Genomes Project (VGP) und der European Reference Genome Atlas (ERGA) – mit der Galaxy-Plattform zusammengeschlossen. Als einer der koordinierenden Partner bietet die Gruppe Environmental Bioinformatics des SIB ihr Fachwissen in den Bereichen Best Practice-Entwicklung, Bereitstellung von Recheninfrastruktur, Verbreitung und Training an.
Über das Galaxy-Framework
Galaxy ist eine quelloffene, webbasierte Plattform für die Analyse biologischer Daten, mit der Benutzer komplexe Arbeitsabläufe für Tausende von Datensätzen und Terabytes an Daten entweder über eine grafische Benutzeroberfläche oder programmgesteuert über Skripte der Anwendungsprogrammierschnittstelle ausführen können. Die wichtigsten globalen Galaxy-Instanzen in den Vereinigten Staaten, der Europäischen Union und Australien sind für Forscher weltweit frei zugänglich und werden durch öffentliche Cloud-Infrastrukturen unterstützt, sodass Benutzer keine Tools installieren oder Infrastruktur beschaffen müssen.
Eine Pipeline zur Demokratisierung der Biodiversitätsgenomik
Forscher haben sich zusammengeschlossen, um innerhalb des Galaxy-Ökosystems – einer Open-Source-Plattform für die FAIR-Datenanalyse – eine digitale Pipeline zu entwickeln, mit der nahezu vollständige Referenzgenom-Assemblierungen erstellt werden können. Um den Assemblierungsprozess zu optimieren und die Qualität sicherzustellen, umfasst dieser Bioinformatik-Workflow umfangreiche Qualitätskontrollfunktionen in jedem Schritt. Durch die Integration komplementärer Sequenzierungstechnologien wird die Pipeline in Zukunft noch effektiver bei der Erstellung vollständiger und genauer Referenzgenome für eine Vielzahl von Spezies sein.
Über das Galaxy-Framework
Galaxy ist eine quelloffene, webbasierte Plattform für die Analyse biologischer Daten, mit der Benutzer komplexe Arbeitsabläufe für Tausende von Datensätzen und Terabytes an Daten entweder über eine grafische Benutzeroberfläche oder programmgesteuert über Skripte der Anwendungsprogrammierschnittstelle ausführen können. Die wichtigsten globalen Galaxy-Instanzen in den Vereinigten Staaten, der Europäischen Union und Australien sind für Forscher weltweit frei zugänglich und werden durch öffentliche Cloud-Infrastrukturen unterstützt, sodass Benutzer keine Tools installieren oder Infrastruktur beschaffen müssen.
In einer kürzlich erschienenen Veröffentlichung beschreiben die Autoren den Ansatz als nützlich für das gesamte Spektrum der Benutzerkenntnisse und Analyseszenarien. Zu diesem Zweck haben sie spezielle Tutorials erstellt, die über das Galaxy Training Network-Portal verteilt werden. Diese Tutorials bieten einen detaillierten Überblick über den Assemblierungsprozess und enthalten eine vereinfachte Version, die die sofortige Nutzung der Workflows erleichtern soll.
Für Robert Waterhouse, Direktor der Environmental Bioinformatics-Gruppe von SIB und Vorsitzender von ERGA, hat diese Zusammenarbeit „Bioinformatiker mit Genomik-Experten zusammengebracht, um modernste Workflows für die Genom-Assemblierung und Qualitätskontrolle zu entwickeln und diese Forschern weltweit frei zugänglich zu machen“.
Reference(s)
Larivière, D., Abueg, L., Brajuka, N. et al. Skalierbare, zugängliche und reproduzierbare Referenzgenom-Assemblierung und -Bewertung in Galaxy. Nat Biotechnol 42, 367–370 (2024).
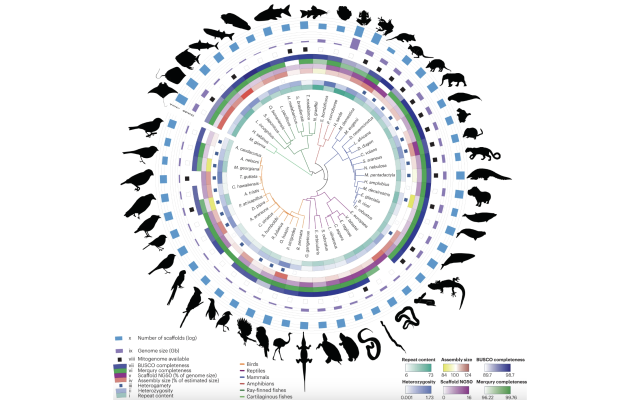
Bildunterschrift: Stammbaum und Assemblierungsstatistik von Genomen, die mit der Galaxy-Assemblierungs-Pipeline assembliert wurden.