Un collectif scientifique, dirigé par le Earth BioGenome Project, vise à séquencer et assembler les génomes de 1,8 million d'espèces animales, végétales et autres eucaryotes d'ici 2035. Cette initiative produira des données librement accessibles, soutenues par des infrastructures cloud publiques et mises à la disposition des chercheurs du monde entier. Ces génomes de référence sont essentiels pour élaborer des stratégies de conservation efficaces et lutter contre le déclin de la biodiversité. Le SIB apporte sa grande expérience en science des données pour garantir le développement de bonnes pratiques, la mise à disposition d'une infrastructure informatique, la diffusion des résultats et la formation.
Séquençage pour mieux préserver la biodiversité
La biodiversité diminue à un rythme alarmant depuis quelques années, principalement en raison des activités humaines. La production du génome de référence d'une espèce permet d'acquérir des connaissances inestimables sur son histoire évolutive, sa diversité génétique et ses adaptations uniques. Ces informations sont essentielles pour élaborer des stratégies de conservation plus efficaces, telles que l'identification d'individus génétiquement pertinents pour les programmes d'élevage, la restauration de la diversité génétique et la compréhension des vulnérabilités aux maladies et aux changements environnementaux.
Le projet Earth BioGenome Project (EBP) vise à séquencer, cataloguer et caractériser les génomes de toute la biodiversité eucaryote de la Terre. Afin d'optimiser les processus d'assemblage du génome et de définir les meilleures pratiques, deux projets génomiques – le Vertebrate Genomes Project (VGP) et l'European Reference Genome Atlas (ERGA) – ont uni leurs forces à celles de la plateforme Galaxy. En tant que partenaire coordinateur, le groupe Environmental Bioinformatics de la SIB apporte son expertise dans le développement des meilleures pratiques, la mise à disposition d'une infrastructure informatique, la diffusion et la formation.
À propos du framework Galaxy
Galaxy est une plateforme open source basée sur le Web destinée à l'analyse de données biologiques qui permet aux utilisateurs d'exécuter des flux de travail complexes sur des milliers d'ensembles de données et des téraoctets de données, soit via une interface utilisateur graphique, soit de manière programmée via des scripts d'interface de programmation d'applications. Les principales instances Galaxy mondiales aux États-Unis, dans l'Union européenne et en Australie sont librement accessibles aux chercheurs du monde entier et prises en charge par des infrastructures cloud publiques, de sorte que les utilisateurs n'ont pas besoin d'installer des outils ni d'acquérir une infrastructure.
Une voie vers la démocratisation de la génomique de la biodiversité
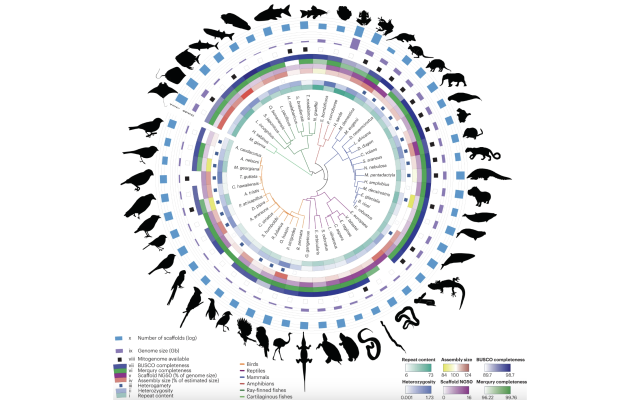
Des chercheurs se sont réunis pour développer un pipeline numérique au sein de l'écosystème Galaxy, une plateforme open source pour l'analyse de données FAIR, afin de générer des assemblages de génomes de référence quasi complets. Afin de rationaliser le processus d'assemblage et d'en garantir la qualité, ce workflow bioinformatique comprend des fonctions de contrôle qualité approfondies à chaque étape. À l'avenir, l'intégration de technologies de séquençage complémentaires rendra le pipeline encore plus efficace pour générer des génomes de référence complets et précis pour une grande variété d'espèces.
À propos du framework Galaxy
Galaxy est une plateforme open source basée sur le Web destinée à l'analyse de données biologiques qui permet aux utilisateurs d'exécuter des flux de travail complexes sur des milliers d'ensembles de données et des téraoctets de données, soit via une interface utilisateur graphique, soit de manière programmée via des scripts d'interface de programmation d'applications. Les principales instances Galaxy mondiales aux États-Unis, dans l'Union européenne et en Australie sont librement accessibles aux chercheurs du monde entier et prises en charge par des infrastructures cloud publiques, de sorte que les utilisateurs n'ont pas besoin d'installer des outils ni d'acquérir une infrastructure.
Dans une publication récente, les auteurs décrivent cette approche comme étant conçue pour être utile à tous les niveaux de compétence des utilisateurs et dans tous les scénarios d'analyse. À cette fin, ils ont créé des tutoriels spécialisés distribués via le portail Galaxy Training Network. Ces tutoriels offrent un aperçu détaillé du processus d'assemblage et comprennent une version simplifiée conçue pour faciliter l'utilisation immédiate des workflows.
Pour Robert Waterhouse, directeur du groupe Environmental Bioinformatics du SIB et président de l'ERGA, cette collaboration « a réuni des bioinformaticiens et des experts en génomique afin de mettre au point des flux de travail de pointe pour l'assemblage et le contrôle qualité des génomes et de les rendre librement accessibles aux chercheurs du monde entier ».
Reference(s)
Larivière, D., Abueg, L., Brajuka, N. et al. Assemblage et évaluation d'un génome de référence évolutif, accessible et reproductible dans Galaxy. Nat Biotechnol 42, 367–370 (2024).
Légende de l'image : Arbre phylogénétique et statistiques d'assemblage des génomes assemblés à l'aide du pipeline d'assemblage Galaxy.