Gli scienziati al SIB, dell'Università di Basilea e della Chelonia Applied Sciences (Basilea) partecipano a un progetto europeo volto a individuare molecole attive contro il COVID-19 e ad accelerare questo processo in futuro. Fanno parte di Exscalate4CoV (E4C), un consorzio pubblico-privato composto da 18 organizzazioni leader dell'UE - tra cui la piattaforma di supercalcolo europea EXSCALATE - provenienti da 7 paesi, che ha ricevuto 3 milioni di euro dal programma Horizon 2020 della Commissione Europea. Gli scienziati utilizzeranno la risorsa del SIB SWISS-MODEL per generare modelli 3D di proteine che non sono ancora state studiate sperimentalmente, accelerando così lo screening virtuale di potenziali farmaci.
Accelerare la ricerca di farmaci contro il COVID-19
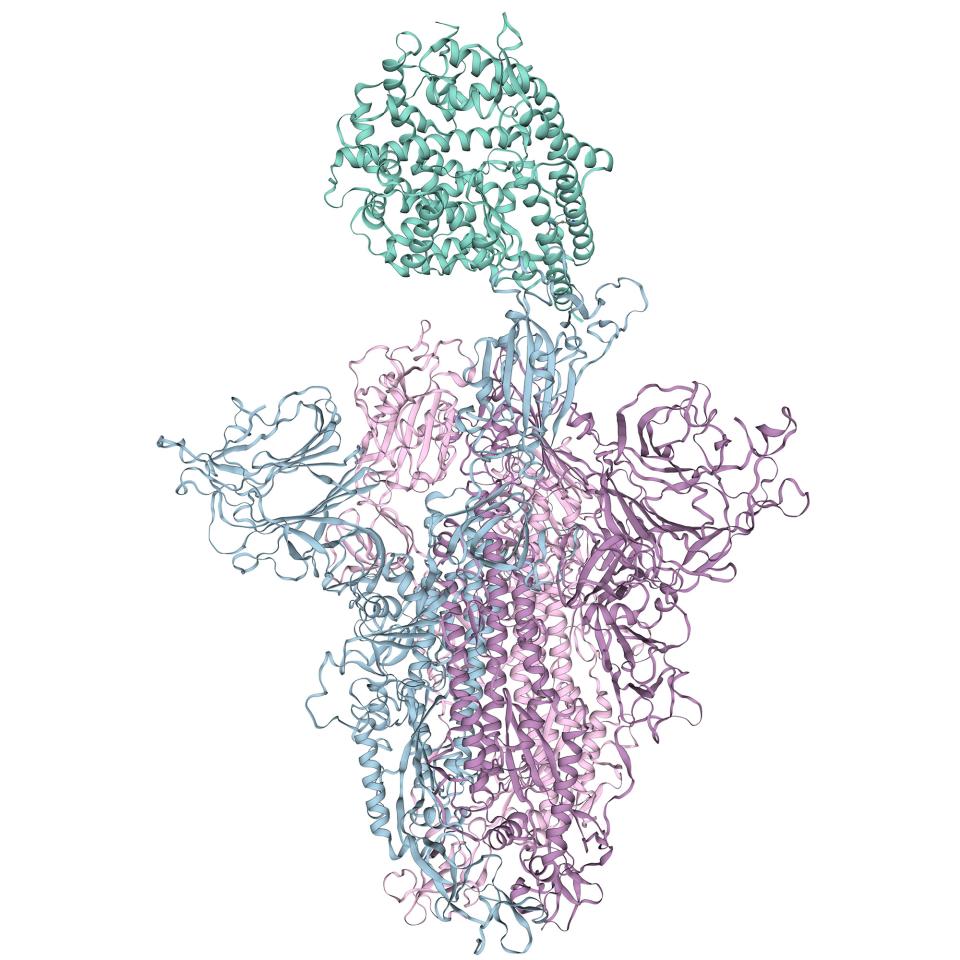
Come parte del consorzio E4C, il gruppo Il SIB Computational Structural Biology (CSB) del Biozentrum (Università di Basilea) è incaricato di generare modelli tridimensionali previsti delle proteine COVID-19 in silico. «Trattandosi di un virus relativamente nuovo, gli scienziati hanno iniziato solo di recente a caratterizzare sperimentalmente le strutture 3D delle proteine virali. I metodi computazionali possono modellare le strutture utilizzando informazioni provenienti da proteine correlate, come quelle del SARS-CoV, accelerando così il processo di scoperta dei farmaci", afferma Gerardo Tauriello, responsabile del team di sviluppo presso il CSB. Questo approccio è chiamato modellazione per omologia e uno degli strumenti principali per eseguirlo è stato sviluppato dal team: il server SWISS-MODEL, una risorsa SIB ad accesso libero. Sia le strutture sperimentali che i modelli di omologia saranno utilizzati dagli altri partecipanti all'E4C per identificare piccole molecole potenzialmente in grado di colpire il coronavirus. Queste molecole saranno poi sottoposte a ulteriori test in esperimentiin vitro, modelli animali e, in caso di successo, sperimentazioni cliniche.
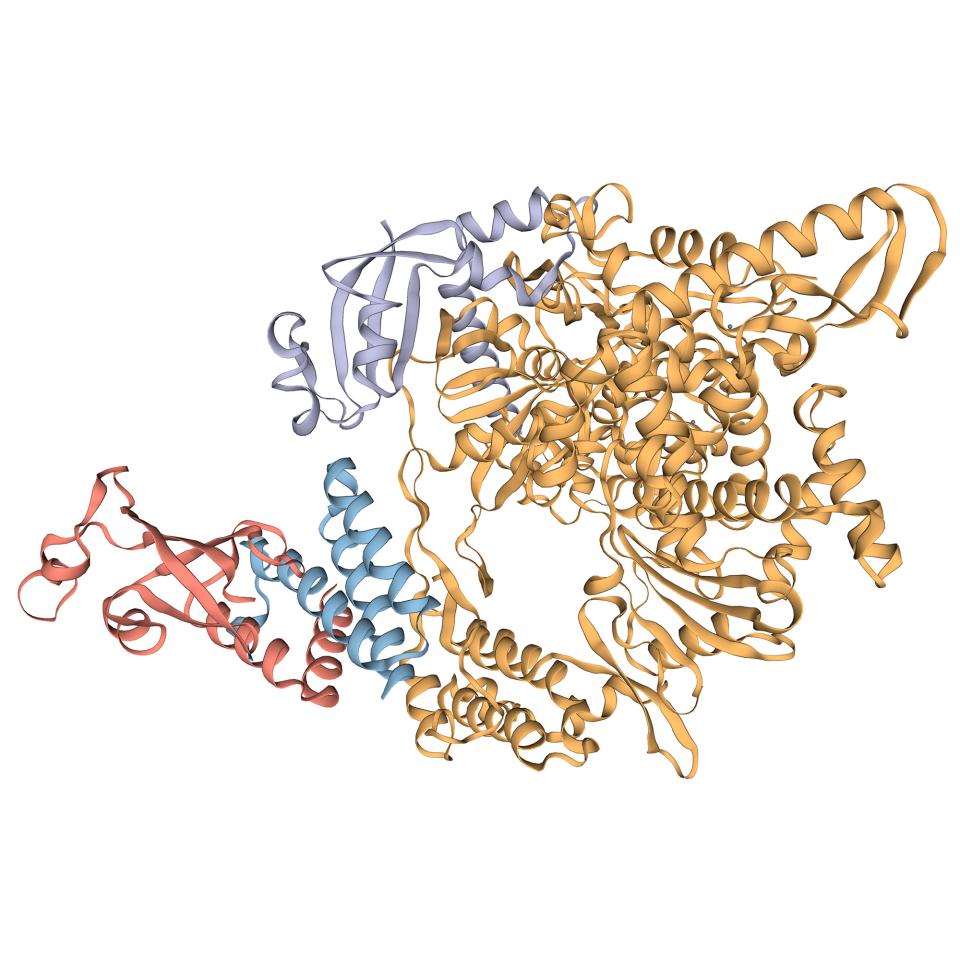
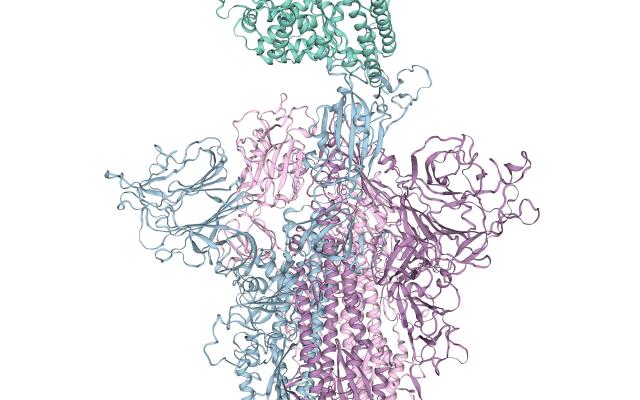
Analisi approfondita delle proteine in 3D per favorire la scoperta di nuovi farmaci
I virus, anche se hanno bisogno delle cellule ospiti per esprimere il loro genoma, dipendono dalle proteine codificate nel loro genoma, proprio come gli organismi viventi. Ogni proteina ha una particolare struttura tridimensionale, strettamente legata alla sua funzione. Ad esempio, una serie di proteine transmembranarie viene utilizzata dal virus per legarsi alle cellule ospiti. Le molecole dei farmaci sono progettate per legarsi alle parti rilevanti di una proteina al fine di modularne la funzione. Una rappresentazione tridimensionale della struttura di una proteina a livello atomico è quindi un aspetto fondamentale nella scoperta razionale di farmaci.
Oltre il COVID-19: accelerare la scoperta di farmaci per future pandemie
E4C mira a sfruttare le conoscenze e gli sviluppi acquisiti durante l'attuale epidemia per accelerare la scoperta e l'identificazione di farmaci in caso di future pandemie, in particolare quando l'agente patogeno è nuovo per la scienza e non esistono trattamenti. «Oggi, l'elaborazione di un nuovo genoma virale richiede un lavoro manuale che richiede molto tempo da parte di esperti di modellizzazione, il che limita i tempi di reazione. In futuro, non appena un nuovo virus sarà sequenziato, qualsiasi ricercatore potrà utilizzare SWISS-MODEL per generare modelli in pochi minuti e averli pronti per l'uso e l'interpretazione all'interno della piattaforma EXSCALATE", spiega Tauriello. Nell'ambito di E4C, il gruppo SIB CSB svilupperà e valuterà un flusso di lavoro automatizzato e scalabile per consentire la generazione di modelli proteici per omologia, direttamente dal genoma di qualsiasi virus.
Collegare i migliori laboratori di ricerca nel campo delle scienze della vita con potenti supercomputer
E4C sta collegando la piattaforma europea di supercalcolo EXSCALATE (EXaSCale smArt pLatform Against paThogEns) - in grado di sfruttare una libreria di 500 miliardi di farmaci candidati, grazie a una capacità di elaborazione di oltre 3 milioni di molecole al secondo - con i migliori laboratori di ricerca nel campo delle scienze della vita. Chelonia Applied Science, con sede a Basilea, ha progettato il consorzio E4C ed è responsabile delle sue attività di diffusione e valorizzazione. Silvano Coletti, amministratore delegato di Chelonia, afferma: "La situazione che il mondo sta affrontando con la pandemia di COVID-19 non ha precedenti nella nostra vita e ha un impatto su tutti noi, sia a livello professionale che personale. Exscalate4CoV è uno sforzo computazionale altrettanto senza precedenti, che mira ad accelerare l'identificazione dei farmaci contro il COVID-19 oggi e a diventare la piattaforma di riferimento per le future pandemie domani, compreso il processo di scoperta e sviluppo nella ricerca farmaceutica".
Oltre ai membri svizzeri, il consorzio E4C, coordinato dall'azienda farmaceutica italiana Domp, comprende 16 istituzioni* provenienti da sei paesi. Per saperne di più, visitate il sito web EXSCALATE4CoV.
*Dompé Farmaceutici SPA, Politecnico di Milano (Dipartimento di Elettronica, Informatica e Bioingegneria), Consorzio Interuniversitario CINECA (Innovazione e Applicazioni Supercomputing), Università degli Studi di Milano (Dipartimento di Scienze Farmaceutiche), Katholieke Universiteit Leuven, International Institute Of Molecular And Cell Biology In Warsaw (LIMCB), Elettra Sincrotrone Trieste, Fraunhofer Institute for Molecular Biology and Applied Ecology, Bsc Supercomputing Centre, Forschungszentrum Jülich, Università Federico II di Napoli, Università degli Studi di Cagliari, KTH Royal Institute of Technology (Dipartimento di Fisica Applicata), Associazione BigData, Istituto Nazionale di Fisica Nucleare (INFN) e Istituto nazionale per le malattie infettive Lazzaro Spallanzani.