Un team guidato da Joana Pereira e Torsten Schwede presso il Biozentrum dell'Università di Basilea e al SIB ha scoperto un tesoro di proteine non caratterizzate. Sfruttando la recente rivoluzione del deep learning e beneficiando di una sovvenzione Kickstarter dedicata da parte di Il SIB, hanno scoperto centinaia di nuove famiglie di proteine, tra cui una nuova struttura proteica prevista. Lo studio è stato ora pubblicato sulla rivista Nature, insieme a un altro studio del gruppo di Pedro Beltrao presso l'ETH su un argomento correlato.
Negli ultimi anni, AlphaFold ha rivoluzionato la scienza delle proteine. Questo strumento di intelligenza artificiale (AI) è stato addestrato sui dati relativi alle proteine raccolti dagli scienziati nel corso di oltre 50 anni ed è in grado di prevedere la forma tridimensionale delle proteine con elevata precisione. Il suo successo ha portato alla modellizzazione di ben 215 milioni di proteine lo scorso anno, fornendo informazioni dettagliate sulla forma di quasi tutte le proteine. Ciò è particolarmente interessante per le proteine che non sono state studiate sperimentalmente, un processo complesso e dispendioso in termini di tempo.
"Ora ci sono molte fonti di informazioni sulle proteine, che racchiudono preziose intuizioni su come le proteine si evolvono e funzionano", afferma Joana Pereira, responsabile dello studio. Tuttavia, la ricerca si è trovata a lungo di fronte a una giungla di dati. Il team di ricerca guidato da Torsten Schwede, responsabile del gruppo presso il Biozentrum dell'Università di Basilea, è ora riuscito a decifrare alcune delle informazioni nascoste.
Una visione dall'alto rivela nuove famiglie e pieghe proteiche
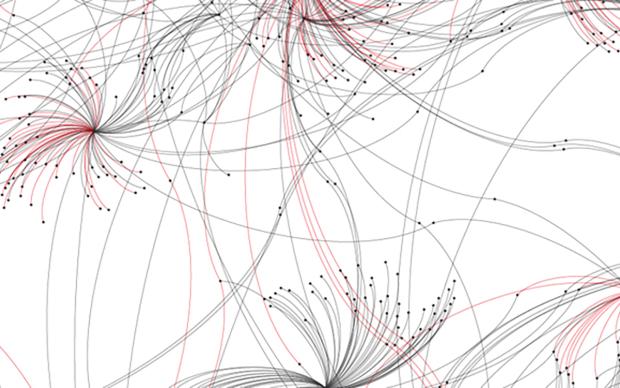
I ricercatori hanno costruito una rete interattiva di 53 milioni di proteine con strutture AlphaFold di alta qualità. «Questa rete costituisce una preziosa fonte per prevedere teoricamente famiglie proteiche sconosciute e le loro funzioni su larga scala», sottolinea Janani Durairaj, primo autore dello studio. Il team è riuscito a identificare 290 nuove famiglie proteiche e una nuova struttura proteica che ricorda la forma di un fiore.
Basandosi sull'esperienza del gruppo Schwede nello sviluppo e nella manutenzione del software leader SWISS-MODEL, supportato da Il SIB, hanno reso la rete disponibile come risorsa web interattiva, denominata "Protein Universe Atlas".
Maggiori informazioni sul nostro portafoglio di software e database leader
Uno dei due articoli rivoluzionari del SIB Groups per esplorare la materia oscura proteica
Il documento qui descritto è uno dei due articoli condotti dai gruppi al SIB che saranno pubblicati sull'argomento. Utilizzando un approccio complementare, basato sulla struttura delle proteine piuttosto che sulle sequenze, il team di Pedro Beltrao dell'ETH di Zurigo e il team di Martin Steinegger e dell'Università Nazionale di Seul propongono un metodo efficiente per raggruppare le proteine presenti nel database AlphaFold. Il risultato principale è stato quello di scoprire quanti tipi diversi di forme proteiche esistono, quanti di questi sono poco studiati e capire quando potrebbero essere comparsi nel corso dell'evoluzione. Entrambi gli articoli sono stati citati in un'intervista pubblicata su Nature.
L'intelligenza artificiale come strumento prezioso nella ricerca
Il team ha utilizzato strumenti basati sul deep learning per individuare novità in questa rete, aprendo la strada a innovazioni nel campo delle scienze della vita, dalla ricerca di base a quella applicata. "Comprendere la struttura e la funzione delle proteine è in genere uno dei primi passi per sviluppare un nuovo farmaco o modificarne le funzioni tramite l'ingegneria proteica, ad esempio", afferma Pereira. Il lavoro è stato sostenuto da una sovvenzione "kickstarter" del SIB per incoraggiare l'adozione dell'IA nelle risorse delle scienze della vita. Ciò sottolinea il potenziale trasformativo del deep learning e degli algoritmi intelligenti nella ricerca.
Con il Protein Universe Atlas, gli scienziati possono ora approfondire le loro conoscenze sulle proteine rilevanti per la loro ricerca. "Ci auguriamo che questa risorsa sia di aiuto non solo ai ricercatori e ai biocuratori, ma anche agli studenti e agli insegnanti, fornendo una nuova piattaforma per l'apprendimento della diversità delle proteine, dalla struttura alla funzione, fino all'evoluzione", afferma Durairaj.
La rete interattiva è accessibile gratuitamente in tutto il mondo qui.
Reference(s)
Durairaj J et al. Alla scoperta di nuove famiglie e pieghe nell'universo delle proteine naturali. Nature. Pubblicato online il 13 settembre 2023.
Barrio-Hernandez I et al. Strutture previste dal clustering su scala dell'universo proteico conosciuto. Nature. Pubblicato online il 13 settembre 2023.