Le innovazioni evolutive degli insetti e degli altri artropodi sono tanto numerose quanto meravigliose, dalle terrificanti zanne e pungiglioni alle ali dai colori raffinati e alle ingegnose prodezze ingegneristiche. Il sequenziamento del DNA ci permette di tracciare i modelli genomici alla base di questa incredibile diversità che caratterizza gli artropodi e li rende il gruppo di animali di maggior successo sulla Terra. Un team internazionale di scienziati riporta sulla rivista Genome Biology i risultati di un progetto pilota, co-diretto dal leader del gruppo Il SIB Robert Waterhouse dell'Università di Losanna, per avviare l'iniziativa globale di sequenziamento di migliaia di artropodi. Analisi comparative su 76 specie che abbracciano 500 milioni di anni di evoluzione rivelano cambiamenti genomici dinamici che indicano i fattori chiave alla base del loro successo e aprono molte nuove aree di ricerca.
Amici e nemici, gli artropodi dominano il mondo
Gli artropodi costituiscono il gruppo di animali più ricco di specie e più diversificato sulla Terra, con numerosi adattamenti nel corso di 500 milioni di anni di evoluzione che hanno permesso loro di sfruttare tutti i principali ecosistemi. Essi svolgono un ruolo fondamentale nell'equilibrio ecologico del nostro pianeta, oltre ad essere sia benefici che dannosi per il successo dell'umanità attraverso l'impollinazione e il riciclaggio dei rifiuti biologici, o la distruzione dei raccolti e la diffusione di malattie. "Sequenziando e confrontando i loro genomi possiamo iniziare a identificare alcuni dei fattori genetici chiave alla base del loro successo evolutivo", spiega Waterhouse, "ma l'impatto delle attività umane nei tempi moderni porrà fine al loro dominio, o la loro capacità di adattarsi e innovarsi garantirà la loro sopravvivenza?"
Il progetto pilota i5k: avvio del sequenziamento del genoma degli artropodi
L'iniziativa i5k, volta a sequenziare e annotare il genoma di 5000 specie di insetti e altri artropodi, è stata lanciata nel 2011 con una lettera pubblicata sulla rivista Science. Fin dall'inizio, l'iniziativa mirava a sostenere lo sviluppo di nuove risorse genomiche per comprendere la biologia molecolare e l'evoluzione degli artropodi. Da allora, l'i5k è cresciuta fino a diventare un'ampia comunità di scienziati che utilizzano la genomica per studiare insetti e altri artropodi in molti contesti diversi, dalla biologia animale fondamentale agli effetti sull'ecologia e l'ambiente, fino all'impatto sulla salute umana e sull'agricoltura. Per dare il via all'iniziativa i5k, è stato avviato un progetto pilota presso il Baylor College of Medicine, guidato da Stephen Richards, per sequenziare, assemblare e annotare i genomi di 28 diverse specie di artropodi accuratamente selezionate tra 787 candidature della comunità.
Confronto su larga scala tra genomi di diverse specie
"L'identificazione e l'annotazione di migliaia di geni dal progetto pilota i5k aumenta notevolmente il nostro attuale campionamento genomico degli artropodi", afferma Waterhouse. Combinando questi dati con genomi precedentemente sequenziati, i ricercatori hanno potuto eseguire un'analisi comparativa su larga scala su 76 specie diverse, tra cui mosche, farfalle, falene, coleotteri, api, formiche, vespe, insetti veri e propri, tripidi, pidocchi, scarafaggi, termiti, effimere, libellule, damigelle, bristletails, crostacei, millepiedi, ragni, zecche, acari e scorpioni. I dottorandi Gregg Thomas dell'Università dell'Indiana, USA, ed Elias Dohmen dell'Università di Münster, Germania, hanno utilizzato i genomi annotati per eseguire analisi evolutive computazionali su oltre un milione di geni di artropodi
L'evoluzione dinamica delle famiglie geniche: la chiave del successo?
Le analisi del team si sono concentrate sul tracciamento delle storie evolutive dei geni per stimare i cambiamenti nel contenuto e nella struttura dei geni nel corso di 500 milioni di anni. Ciò ha consentito di identificare famiglie di geni che hanno subito un aumento o una diminuzione sostanziale delle dimensioni, o che sono emersi o scomparsi di recente, o che hanno riorganizzato i loro domini proteici, tra e all'interno di ciascuno dei principali sottogruppi di artropodi. Le famiglie di geni che hanno subito i cambiamenti più dinamici codificano proteine coinvolte in funzioni legate alla digestione, alla difesa chimica e alla costruzione e al rimodellamento della chitina, una parte importante dell'esoscheletro degli artropodi. L'adattabilità dei processi digestivi e dei meccanismi di neutralizzazione delle sostanze chimiche nocive ha indubbiamente aiutato gli artropodi a conquistare un'ampia varietà di nicchie ecologiche. Forse ancora più importante, la flessibilità derivante da un corpo segmentato e da un esoscheletro dinamicamente rimodellabile ha permesso loro di prosperare adattandosi fisicamente ai nuovi ecosistemi.

Innovazione attraverso l'invenzione e il riutilizzo
Le famiglie di geni di recente evoluzione riflettono anche funzioni note per essere importanti in diversi gruppi di artropodi, come l'apprendimento visivo e il comportamento, il rilevamento di feromoni e odori, l'attività neuronale e lo sviluppo delle ali. Queste possono migliorare le capacità di localizzazione del cibo o perfezionare l'auto-riconoscimento e la comunicazione delle specie. Al contrario, sono stati identificati pochi cambiamenti nell'antenato degli insetti che subiscono una metamorfosi completa: il cambiamento radicale dalla forma giovanile all'adulto completamente sviluppato (come un bruco che si trasforma in farfalla). Questo è stato tradizionalmente considerato un passo importante nell'evoluzione degli insetti dallo stato originale di sviluppo attraverso fasi ninfali graduali fino al raggiungimento dello stadio adulto. "Questi risultati supportano l'idea che questa transizione chiave sia avvenuta più probabilmente attraverso il ricablaggio delle reti geniche esistenti o la costruzione di nuove reti utilizzando geni esistenti, uno scenario di nuovi trucchi per vecchi geni", spiega Waterhouse.
Delineazione ortologica con OrthoDB: una risorsa al SIB di SwissOrthology
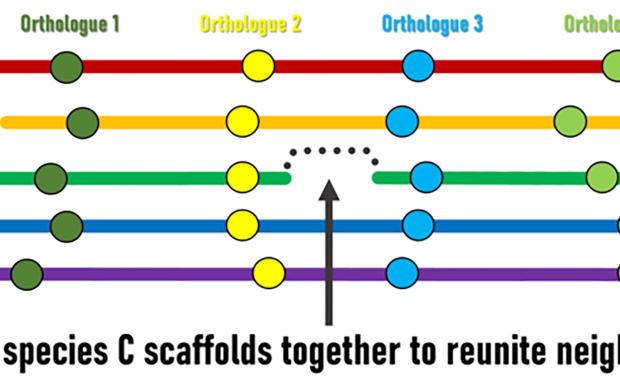
La delineazione ortologica, ovvero l'identificazione di geni "equivalenti" provenienti da specie diverse, è un pilastro della genomica comparativa. Consente di tracciare la storia evolutiva di migliaia di geni provenienti da molte specie e offre ipotesi qualificate sulle presunte funzioni dei geni. Gli ortologhi degli artropodi sono stati identificati utilizzando la procedura di delineazione dell'ortologia OrthoDB, una risorsaSwissOrthology SIB. Per trovare gli ortologhi, le sequenze proteiche dei geni di tutte le specie incluse vengono prima confrontate per identificare insiemi di geni simili. Gli algoritmi di clustering basati su grafici creano quindi gruppi di ortologhi che rappresentano tutti i discendenti di un singolo gene presente nel genoma dell'ultimo antenato comune di tutte le specie. Il numero crescente di genomi sequenziati significa che tali metodi comparativi stanno diventando strumenti sempre più potenti per migliorare sia le annotazioni strutturali dei geni a livello genomico sia le inferenze funzionali dei geni su larga scala. Questi approcci sono ormai consolidati e considerati estremamente preziosi per la scoperta e la caratterizzazione dei geni, contribuendo a creare risorse a supporto della ricerca biologica.
Approfondimenti genomici sulla biologia e l'evoluzione degli artropodi
Diversi studi genomici dettagliati su singole specie i5k si sono concentrati sulle loro affascinanti caratteristiche biologiche, quali l'ecologia alimentare e la biologia dello sviluppo della cimice del latte, la resistenza agli insetticidi, l'alimentazione ematica e il sesso traumatico della cimice dei letti, il trasferimento genico orizzontale da batteri e funghi e la digestione di materiali vegetali da parte del coleottero asiatico dal lungo corno, nonché le interazioni parassita-ospite e i potenziali vaccini per la mosca della pecora. Le analisi combinate rivelano famiglie di geni in continua evoluzione e di nuova comparsa che stimoleranno nuovi ambiti di ricerca. "Possiamo portare queste ipotesi in laboratorio e utilizzarle per studiare direttamente come il genoma si traduce in morfologia visibile con una risoluzione che non può essere raggiunta con nessun altro gruppo animale", afferma il coautore Ariel Chipman, dell'Università Ebraica di Gerusalemme, Israele. Le nuove risorse fanno progredire in modo sostanziale la creazione di un catalogo genomico completo della vita sul nostro pianeta e, con oltre un milione di specie di artropodi descritte e una stima di altre sette volte tanto, c'è chiaramente ancora molto da scoprire!
I prossimi passi nella genomica degli artropodi e oltre
Grazie a tecnologie di sequenziamento del DNA più efficaci ed economiche, sono già in corso nuove ambiziose iniziative volte a sequenziare il genoma di altri artropodi. Tra queste figurano la Global Ant Genome Alliance e la Global Invertebrate Genomics Alliance, nonché il Darwin Tree of Life Project, che ha come obiettivo tutte le specie animali conosciute delle isole britanniche, e la rete globale di comunità coordinate dall'Earth BioGenome Project (EBP), che mira a sequenziare tutta la biodiversità eucariotica della Terra7. Gli obiettivi dell'EBP includono anche il benessere umano, dove il ruolo degli artropodi è chiaro e i benefici nascosti sono probabilmente sostanziali, nonché la protezione della biodiversità e la comprensione degli ecosistemi, dove i rapporti allarmanti sul calo del numero di artropodi rendono questi ultimi una priorità. "Il completamento del progetto pilotai5k rappresenta quindi una tappa importante nel progresso verso l'intensificazione degli sforzi per sviluppare un catalogo genomico completo della vita sul nostro pianeta", conclude Richards.
Reference(s)
Evoluzione del contenuto genetico negli artropodi. Thomas et al., 2020, Genome Biology.