L'intelligence artificielle a remporté ses premiers prix Nobel, notamment pour des outils révolutionnaires dans le domaine des sciences de la vie, et l'IA générative est de plus en plus puissante et répandue. Le SIB catalyse, facilite et exploite ces avancées transformatrices dans le domaine de l'IA, parmi d'autres.
L'expertise et les données du SIB sont essentielles au modèle d'IA récompensé par le prix Nobel
AlphaFold a été récompensé par le prix Nobel de chimie 2024 pour sa capacité à prédire les structures protéiques en 3D à partir de leur séquence d'acides aminés. Le modèle s'appuie sur plusieurs décennies d'expertise en bio-informatique, notamment trois ressources ouvertes et initiatives développées et co-développées par les scientifiques du SIB :
- Le modèle d'IA a appris à identifier les relations entre les séquences d'acides aminés et les structures 3D en analysant des centaines de millions de séquences protéiques de haute qualité dans UniProt. Les annotations d'experts sur la structure des protéines ont également aidé les développeurs d'AlphaFold à comprendre et à déboguer les performances du modèle.
- Son impressionnante précision a d'abord été démontrée par le CASP, un concours mondial organisé tous les deux ans qui teste la précision des méthodes computationnelles par rapport à des structures protéiques déterminées expérimentalement, non publiées et très complexes. Torsten Schwede, chef de groupe au SIB, est membre du comité organisateur du CASP depuis 2011.
- L'applicabilité d'AlphaFold à toutes les protéines humaines a ensuite été confirmée à l'aide des données de CAMEO, qui effectue le même test de précision que le CASP, mais sur un ensemble plus large de protéines publiées chaque semaine.
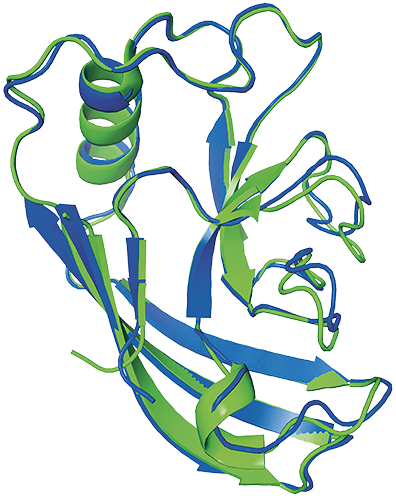
Structure protéique prédite par AlphaFold (en bleu) et expérimentalement (en vert)
Fournir des données de formation de référence grâce à des bases de données organisées
Les bases de données organisées par le SIB fournissent des données et des connaissances hautement fiables à partir desquelles les modèles d'IA peuvent apprendre à reconnaître des modèles et à faire des prédictions pertinentes. L'une d'entre elles, UniProt, a joué un rôle crucial dans la formation d'AlphaFold (voir encadré). De nombreuses autres sont disponibles et utilisées dans des applications d'IA pour relever des défis complexes dans le domaine des sciences de la vie.
L'expertise et les données du SIB sont essentielles au modèle d'IA récompensé par le prix Nobel
AlphaFold a été récompensé par le prix Nobel de chimie 2024 pour sa capacité à prédire les structures protéiques en 3D à partir de leur séquence d'acides aminés. Le modèle s'appuie sur plusieurs décennies d'expertise en bio-informatique, notamment trois ressources ouvertes et initiatives développées et co-développées par les scientifiques du SIB :
- Le modèle d'IA a appris à identifier les relations entre les séquences d'acides aminés et les structures 3D en analysant des centaines de millions de séquences protéiques de haute qualité dans UniProt. Les annotations d'experts sur la structure des protéines ont également aidé les développeurs d'AlphaFold à comprendre et à déboguer les performances du modèle.
- Son impressionnante précision a d'abord été démontrée par le CASP, un concours mondial organisé tous les deux ans qui teste la précision des méthodes computationnelles par rapport à des structures protéiques déterminées expérimentalement, non publiées et très complexes. Torsten Schwede, chef de groupe au SIB, est membre du comité organisateur du CASP depuis 2011.
- L'applicabilité d'AlphaFold à toutes les protéines humaines a ensuite été confirmée à l'aide des données de CAMEO, qui effectue le même test de précision que le CASP, mais sur un ensemble plus large de protéines publiées chaque semaine.

Structure protéique prédite par AlphaFold (en bleu) et expérimentalement (en vert)
- Transformer les données en connaissances prêtes pour l'IA
Nos bases de données sont ouvertes, FAIR et lisibles par machine, grâce à des données harmonisées, des métadonnées riches et un contrôle qualité rigoureux.
Les bases de données organisées par des experts comprennent en outre des informations pertinentes et continuellement mises à jour provenant de la littérature scientifique et d'autres sources. Nos biocurateurs annotent les séquences protéiques dans UniProt, par exemple, avec des connaissances sur la structure, la fonction et d'autres aspects des protéines.
Ce codage explicite d'informations complexes dans des formats lisibles par machine est essentiel pour les progrès de l'IA dans le domaine scientifique.- Une mine de données d'entraînement librement accessibles
Le SIB co-développe des bases de données ouvertes et organisées couvrant les biomolécules et les processus de l'arbre de la vie.
On peut citer par exemple l'expression génétique (Bgee), les lignées cellulaires (Cellosaurus), les glucides (Glyco@Expasy), les réactions (Rhea), les interactions protéiques (STRING), les lipides (SwissLipids), les génomes pathogènes (SPSP), les orthologues (SwissOrthology), les séquences et structures protéiques (UniProt, Swiss-Model) et les fonctions génétiques (Gene Ontology).
- Une mine de données d'entraînement librement accessibles
- Permettre la formation sur des données sensibles
Nous développons également des méthodes éthiques et sécurisées pour l'analyse par l'IA de données humaines qui ne peuvent être partagées ouvertement. L'analyse fédérée, par exemple, permet d'accéder aux données pertinentes dans un environnement sécurisé sans partager aucune information personnelle.
Les scientifiques du SIB adoptent cette approche dans plusieurs initiatives, notamment l'initiative internationale iCARE4CVD pour la prévention et le traitement personnalisés des maladies cardiovasculaires (en savoir plus) et BioMedIT, l'environnement de recherche fiable du Réseau suisse de santé personnalisée (SPHN; en savoir plus).
Permettre des connaissances exploitables grâce à la représentation des connaissances
Une base de données unique est beaucoup plus puissante lorsque les connaissances qu'elle contient sont reliées à d'autres bases de données. Les scientifiques de la SIB relient les silos de données entre les domaines et les pays en développant et en mettant en œuvre des systèmes permettant de représenter formellement les connaissances, tels que des vocabulaires normalisés pour représenter les entités biologiques et leurs relations (par exemple, les gènes, les protéines, les métabolites, les espèces, les maladies).
Cela permet à l'IA et aux chercheurs de récupérer et de traiter efficacement des données provenant de diverses sources, de mettre au jour les liens entre ces données et d'obtenir ainsi une vue d'ensemble intégrée des systèmes biologiques complexes.
Voici quelques exemples :
- le développement de la découverte basée sur l'IA dans l'ensemble des résultats de la recherche européenne, grâce au projet Data Commons de l'European Open Science Cloud (EOSC) (en savoir plus) ;
- la mise en place d'une oncologie de précision basée sur l'IA en convertissant des directives PDF non structurées pour le traitement du cancer en procédures de traitement structurées et interopérables, et en créant un outil d'IA pour analyser les procédures et les données cliniques historiques afin de prédire le meilleur traitement pour chaque patient, dans le cadre du projet AI Tumor Board mené avec des hôpitaux suisses ;
- la promotion de la recherche assistée par l'IA dans les connaissances botaniques organisées, en reliant les données chimiques sur les plantes à des informations telles que les interactions entre les espèces et leurs caractéristiques, dans le cadre de l'initiative Digital Botanical Gardens.
Pour en savoir plus sur la représentation des connaissances, consultez le site SIB.
Garantir la fiabilité des résultats grâce à l'analyse comparative
Comment les développeurs d'IA – et les utilisateurs de leurs modèles – peuvent-ils avoir confiance dans les prédictions de l'IA ? Et comment les chercheurs peuvent-ils savoir quel outil d'IA est le mieux adapté à une tâche particulière ? L'expertise de SIB en matière d'analyse comparative a permis de répondre à ces questions pour AlphaFold (voir encadré ci-dessus) et permet une évaluation rigoureuse et un ajustement précis de nombreuses autres analyses informatiques et d'IA.
Nos scientifiques :
- fournissent des ensembles de données de référence de référence qui servent d'entrées standardisées pour comparer les outils d'IA, de résultats cibles par rapport auxquels les prédictions peuvent être évaluées, et de ressources pour affiner les modèles pour des domaines spécifiques ou évaluer la qualité des données avant les analyses ;
- développent des outils logiciels de benchmarking pour évaluer les performances de l'IA par rapport aux données de référence ou à d'autres modèles, mettre en évidence les domaines à améliorer et aider les chercheurs à sélectionner le modèle le mieux adapté à leurs besoins.
Parmi les exemples, on peut citer les outils et les ensembles de données permettant d'évaluer les prédictions relatives au repliement et à l'interaction des protéines (CAMEO et CASP ; voir encadré ci-dessus), la qualité du protéome (SwissOrthology) et la qualité du génome et du métagénome (BUSCO et LEMMI) ; un ensemble de données permettant d'affiner les modèles d'IA générative (voir ci-dessous) pour la curation de données spécialisées (EnzChemRED) ; et un système qui garantit une évaluation comparative reproductible et neutre des outils de biologie computationnelle selon les principes FAIR (Findable, Accessible, Interoperable and Reusable) (Omnibenchmark).
De l'IA générative et des LLM
L'IA générative englobe les systèmes capables de créer de nouveaux contenus, qu'il s'agisse de textes, d'images, de vidéos, de musique ou bien d'autres choses encore. Les grands modèles linguistiques (LLM), un type clé d'IA générative, sont entraînés à partir de données textuelles exhaustives, notamment des séquences génétiques ou du code informatique, afin de résumer, générer et prédire de nouveaux contenus. Des modèles tels que ChatGPT et BioBERT en sont des exemples, ChatGPT excellant dans la génération de texte pour les chatbots et l'écriture créative, tandis que BioBERT se concentre sur (c'est-à-dire est pré-entraîné sur) le texte biomédical. Les LLM utilisent des techniques d'apprentissage profond, en particulier des transformateurs, pour analyser et comprendre les modèles linguistiques à partir de vastes ensembles de données, et pour prédire le prochain « mot » ou la prochaine séquence de mots en fonction du contexte.
Accélérer les découvertes dans le domaine des sciences de la vie grâce à l'IA générative
ChatGPT et une multitude d'autres modèles d'IA générative bouleversent non seulement notre vie quotidienne, mais aussi la science. SIB embrasse cette révolution dans tous les domaines, des applications cliniques à la génération de connaissances biologiques.
De l'IA générative et des LLM
L'IA générative englobe les systèmes capables de créer de nouveaux contenus, qu'il s'agisse de textes, d'images, de vidéos, de musique ou bien d'autres choses encore. Les grands modèles linguistiques (LLM), un type clé d'IA générative, sont entraînés à partir de données textuelles exhaustives, notamment des séquences génétiques ou du code informatique, afin de résumer, générer et prédire de nouveaux contenus. Des modèles tels que ChatGPT et BioBERT en sont des exemples, ChatGPT excellant dans la génération de texte pour les chatbots et l'écriture créative, tandis que BioBERT se concentre sur (c'est-à-dire est pré-entraîné sur) le texte biomédical. Les LLM utilisent des techniques d'apprentissage profond, en particulier des transformateurs, pour analyser et comprendre les modèles linguistiques à partir de vastes ensembles de données, et pour prédire le prochain « mot » ou la prochaine séquence de mots en fonction du contexte.
Voici quelques exemples :
- l'accélération des découvertes biologiques et bioinformatiques grâce à ExpasyGPT, un outil d'IA générative personnalisé intégré à Expasy, le portail suisse de ressources bioinformatiques, qui permet aux chercheurs de récupérer et de compiler des informations provenant des bases de données du SIB plus rapidement et plus facilement que jamais (en savoir plus) ;
- la génération rapide d'anticorps personnalisés pour lutter contre les maladies grâce à AntibodyGPT, qui accélère le processus traditionnellement lent de découverte d'anticorps monoclonaux en prédisant les structures d'anticorps présentant les propriétés souhaitées ;
- tester la capacité de ChatGPT à répondre à des questions médicales dans le domaine de la radiothérapie (lire la publication) ;
- décryptage du rôle caché de l'ARN dans le cancer grâce à l'expertise en traitement du langage naturel ;
- la compréhension du processus de mue des insectes en intégrant les données sur les noms d'espèces et les données de séquences provenant de différentes bases de données publiques à l'aide de méthodes d'IA générative dans la ressource MoultDB, qui sert de référence dans ce domaine.
Découvrez comment les experts du SIB relèvent les défis grâce à l'IA générative.
IA générative et biocuration : un cycle vertueux
L'interaction entre les possibilités offertes par l'IA, et les LLM en particulier, et l'importance de l'expertise humaine est bien illustrée dans le contexte de la biocuration, où le SIB est un leader reconnu. La biocuration est l'art d'extraire de manière experte des connaissances de la littérature biologique et biomédicale pour construire une encyclopédie précise, fiable et à jour au service de la science au sens large.
Dossiers approfondis sur l'IA fiable
- La révolution de l'IA se poursuit – SIB Profile 2025
- IA générative : nouveaux horizons – SIB Profile 2024
- Autour de l'apprentissage automatique – SIB Profile 2021